Содержание
CRISPR/CAS9: что значит для человечества переход от чтения генома к его редактированию?
Как ученые модифицируют гены, какие этические вопросы это может породить и будут ли с помощью генных технологий создавать «детей на заказ»
Технология CRISPR/Cas9, позволяющая вносить изменения в геном высших организмов (в том числе человека) стала в последние годы одной из самых обсуждаемых — не только молекулярными биологами, но и биотех-инвесторами, медиками, социологами — тем. Все дело в том, что CRISPR/Cas9 потенциально имеет перспективы применения для борьбы с многими тяжелыми заболеваниями, среди которых рак, наследственные болезни, ВИЧ. Если раньше генетические технологии применялись в первую очередь для диагностики, то теперь мы впервые подошли к новому рубежу — у нас есть инструмент редактирования ДНК, который, возможно, получит все больше внедрений в клиническую практику и программы лечения. Хотя ранее попытки редактирования генома уже были (например, для больных лейкемией) именно CRISPR/Cas9, как более универсальный инструмент, претендует на создание инструментов для все более активных внедрений. Старт дан: в Китае уже делают первые шаги в клинических испытаниях технологий, основанных на CRISPR/Cas9. Растущие возможности генной терапии ставят перед нами все больше вопросов, связанных с этикой. Чего ждать?
Хотя ранее попытки редактирования генома уже были (например, для больных лейкемией) именно CRISPR/Cas9, как более универсальный инструмент, претендует на создание инструментов для все более активных внедрений. Старт дан: в Китае уже делают первые шаги в клинических испытаниях технологий, основанных на CRISPR/Cas9. Растущие возможности генной терапии ставят перед нами все больше вопросов, связанных с этикой. Чего ждать?
CRISPR/Cas9 — «ножницы» вместо «ножа» для ДНК
Способы редактирования генов в геномах живых существ изучаются с начала XX века – еще с открытия механизма индуцированного мутагенеза (то есть вызванного воздействием каких-нибудь внешних агентов – например, радиоактивного излучения или химических веществ). И если для бактерий механизмы достаточно точного модифицирования генов разработаны еще в середине XX века, то для более сложных организмов, в частности, человека, подходы появились лишь в конце прошлого века. Например, целое семейство вирусов, называемое ретровирусами (к которым относится и ВИЧ, вызывающий СПИД у человека), от природы получило механизм, согласно которому для функционирования вируса требуется встраивание его генома в геном организма-хозяина. Путем введения модификаций в геном ретровируса, то есть вставки измененных человеческих генов, можно добиться внедрения в геном хозяина таких квазичеловеческих элементов – вот и готовый механизм геномного редактирования. Существенным его недостатком является отсутствие специфичности встраивания, то есть вирусный геном может попасть в любой участок генома хозяина, а может и вообще не попасть. Для научных изысканий это нестрашно – всегда можно повторить эксперимент. Но для целей лечения конкретных пациентов подход с «повторить», как правило, не работает.
Например, целое семейство вирусов, называемое ретровирусами (к которым относится и ВИЧ, вызывающий СПИД у человека), от природы получило механизм, согласно которому для функционирования вируса требуется встраивание его генома в геном организма-хозяина. Путем введения модификаций в геном ретровируса, то есть вставки измененных человеческих генов, можно добиться внедрения в геном хозяина таких квазичеловеческих элементов – вот и готовый механизм геномного редактирования. Существенным его недостатком является отсутствие специфичности встраивания, то есть вирусный геном может попасть в любой участок генома хозяина, а может и вообще не попасть. Для научных изысканий это нестрашно – всегда можно повторить эксперимент. Но для целей лечения конкретных пациентов подход с «повторить», как правило, не работает.
Другие способы модификаций генома связаны с технологиями ZFN и TALEN, активно обсуждаемые начиная с 2000-х годов. Идея этих подходов основана также на природных свойствах определенных белков, называемых нуклеазами. Эти активные белки (ферменты) умеют проводить специфическое, неслучайное вырезание участка исходного генома и встраивание в место разреза привнесенного с собой кусочка исправленной ДНК. Такой способ позволяет проводить целевую, гораздо более точную, чем просто ретровирусная, модификацию «сломанных» генов. Отличие ZFN и TALEN заключается в использовании разных видов ферментов, но итог их работы примерно одинаков.
Эти активные белки (ферменты) умеют проводить специфическое, неслучайное вырезание участка исходного генома и встраивание в место разреза привнесенного с собой кусочка исправленной ДНК. Такой способ позволяет проводить целевую, гораздо более точную, чем просто ретровирусная, модификацию «сломанных» генов. Отличие ZFN и TALEN заключается в использовании разных видов ферментов, но итог их работы примерно одинаков.
В 2015 году технологию ZFN удалось успешно применить для терапии ВИЧ: в стволовых клетках донора был отредактирован участок, отвечающий за восприимчивость ВИЧ, затем они были трансплантированы пациенту. Стоит отметить, что примерно у каждого тысячного европейца имеется такой генотип, который гарантирует невозможность внедрения вириона (активной вирусной частицы, которая осуществляет заражение) внутрь клеток организма-хозяина, то есть невозможность инфицирования.
Если разрезать последовательность ДНК в двух местах, то можно воспользоваться естественными механизмами репарации ее цепочки — не дать ей соединиться с другой ДНК или просто «сшить» два конца, а добавить в место разрыва нужный, введенный заранее, фрагмент. Место надреза помогают локализировать специальные белки, которые привязаны к определенным последовательностям генов и связывающие участки генома. Оказалось, что у таких белков есть участки с ионом цинка, которые делают их еще более специфичными фрагменту ДНК — соотносит их только с тремя нуклеотидами. Если прикрепить эти «цинковые пальцы» генно-инженерных белков в качестве идентификаторов к «ножницам» (их роль выполняет специальный вид нуклеазы, фермента для реакций между нуклеиновыми кислотами) — можно достаточно точно выявить последовательности генов с обеих сторон от места нужного «разреза». Так работает технология ZFN (Zinc finger nuclease). В основе технологии TALEN (Transcription Activator-Like Effector Nuclease) — схожие принципы, только для создания ДНК-связывающего участка белка, «знающего» нужную последовательность генов, используются TAL-белки.
Место надреза помогают локализировать специальные белки, которые привязаны к определенным последовательностям генов и связывающие участки генома. Оказалось, что у таких белков есть участки с ионом цинка, которые делают их еще более специфичными фрагменту ДНК — соотносит их только с тремя нуклеотидами. Если прикрепить эти «цинковые пальцы» генно-инженерных белков в качестве идентификаторов к «ножницам» (их роль выполняет специальный вид нуклеазы, фермента для реакций между нуклеиновыми кислотами) — можно достаточно точно выявить последовательности генов с обеих сторон от места нужного «разреза». Так работает технология ZFN (Zinc finger nuclease). В основе технологии TALEN (Transcription Activator-Like Effector Nuclease) — схожие принципы, только для создания ДНК-связывающего участка белка, «знающего» нужную последовательность генов, используются TAL-белки.
Но ZFN и TALEN оказались далеки от массового применения в медицине. Ученые пытались научить их узнавать специфическую, в идеале — любую заданную последовательность ДНК для «кусания». Иногда это работало, но для каждой последовательности приходилось создавать свой отдельный белок, а это кропотливая и долгая работа.
Иногда это работало, но для каждой последовательности приходилось создавать свой отдельный белок, а это кропотливая и долгая работа.
Иное дело — редактирование генома с помощью системы CRISPR/Cas9. В ней используется один белок (комплексы белков CAS), а «путеводитель» для нахождения участка ДНК для необходимых модификаций можно сделать очень быстро и дешево. Благодаря определенным участкам генома CRISPR, которые хранят генетические последовательности вирусов, белок CAS разрезает соответствующие последовательности. С этим могут работать очень и очень многие хорошо оборудованные лаборатории. Именно поэтому технология CRISPR/Cas9, изначально открытая как защитный механизм бактерий от вирусов в конце 1980-х, вызвала такой ажиотаж.
Пока неочевидно, какие технологии геномного редактирования будут наиболее активно применяться в медицине уже через 10 лет. Возможно, это будет CRISPR/Cas9 или текущие аналоги, а может быть, будет открыта новая технология, которая возникнет так же неожиданно и ярко, как CRISPR/Cas9.
А пока между двумя научными группами, которые в 2012 году одновременно нашли способ применения CRISPR/Cas9 для точечного редактирования геномов сложных организмов, идет патентная война. Группа в Калифорнийском университете в Беркли и группа из MIT и Broad Institute (институт MIT и Гарварда), подавшие заявки на патент в разное время в 2013 году, тратят десятки миллионов долларов на юристов и вряд ли остановятся — на кону миллиарды долларов, которые может принести технология. По прогнозам, патентный офис примет решение в 2017 году.
Венчурные инвесторы уже активно инвестируют в компании, развивающие CRISPR/Cas9. Только за 2016 год три такие компании вышли на IPO. Капитализация первой, Editas Medicine, составляет $542 млн, а среди ее инвесторов до выхода на NASDAQ — Билл Гейтс. Вторая, Intellia Therapeutics, достигла капитализации $600 млн, а среди ее первых инвесторов оказался швейцарский фармацевтический гигант Novartis (№ 47 в глобальном списке Forbes). Третья, CRISPR Therapeutics, пока прошла отметку в капитализации лишь в $107 млн, в ее финансировании участвует фармкомпания Bayer (№97 в глобальном списке Forbes).
Третья, CRISPR Therapeutics, пока прошла отметку в капитализации лишь в $107 млн, в ее финансировании участвует фармкомпания Bayer (№97 в глобальном списке Forbes).
Применение и этические вопросы
Среди потенциальных применений новой технологии — лечение наследственных заболеваний (гемофилия, бета-талассемия, мышечная дистрофия), терапия онкологии и вирусных инфекций, включая ВИЧ.
Но есть и более экзотические потенциальные применения. Например, борьба с мультифакторными заболеваниями (диабет, шизофрения и др.) или редактирование эмбрионов при искусственном оплодотворении для подбора определенной внешности у детей. Именно здесь возникает множество этических вопросов, которые начали обсуждаться, но пока так и не получили консенсусного решения у мирового сообщества. Когда же можно, а когда нельзя применять редактирование генома? Пока, в отсутствие у человечества единой позиции, каждая из стран решает это по-своему.
Например, мир потрясли уже два исследования китайских ученых. В 2015 году группа китайских ученых применила геномное редактирование на 86 человеческих эмбрионах для того, чтобы изменить мутации, вызывающие тяжелую наследственную патологию бета-талассемию. Это тяжелая наследственная патология, которая связана с нарушением синтеза гемоглобина и разрушением эритроцитов, средняя продолжительность жизни носителей мутации — 17 лет. Несмотря на серьезную научную значимость исследования китайских ученых, два главных научных журнала Nature (Великобритания) и Science (США) отказались публиковать результаты, в частности из-за этических вопросов. Также это исследование показало неидеальность технологии CRISPR/Cas9, по крайней мере на данный момент. Из 86 эмбрионов точно поменять желаемый участок ДНК получилось только у 28. Процент ошибки оказался больше, чем ожидали исследователи исходя из опытов над эмбрионами животных. Какой участок ДНК нужно редактировать, определяется при помощи синтетической последовательности РНК (так называемый «гид»). Она комплементарна нужному участку ДНК. Но может оказаться, что в другой части генома также есть аналогичная последовательность нуклеотидов.
Она комплементарна нужному участку ДНК. Но может оказаться, что в другой части генома также есть аналогичная последовательность нуклеотидов.
Исследование вызвало множество дискуссий. Должны ли западные страны очень аккуратно относиться к этическим вопросам при применении новых технологий геномного редактирования, либо же это приведет только к отставанию от Китая? По всей видимости, пока Запад рассматривает возможность терпимее относиться к генетическим модификациям — менее чем через полгода после скандальной публикации китайских исследователей в Великобритании были официально разрешены первые опыты по геномному редактированию эмбрионов человека, в Лондоне новые группы ученых уже ведут работу над дизайном новых экспериментов.
А в середине ноября группа китайских ученых анонсировала применение генной модификации клеток при помощи CRISPR/Cas9 для лечения пациента с агрессивным раком легкого. В журнале Nature эта статья анонсирована с подзаголовком: «Шаг китайских ученых может разжечь борьбу между Китаем и США в сфере биомедицины». Мир ждет результатов второго исследования китайских ученых.
Мир ждет результатов второго исследования китайских ученых.
Однако в будущем этических вопросов, связанных с новыми технологиями в области генетики и репродукции, будет еще больше. Биоэтика становится все более важной дисциплиной.
Например, у некоторых европейцев есть мутации в гене CCR5 — ее носители практически невосприимчивы к ВИЧ. В рамках генетического тестирования эти мутации могут быть исследованы. Но этично, корректно ли рассказывать человеку о наличии такой мутации? Мы, как компания, которая занимается генетическими тестами, решили, что нет.
Другие проблемы связаны с тем, что возможности генетического редактирования меняют само понятие семьи. С появлением искусственного оплодотворения и суррогатного материнства в принципе понимание института семьи усложнилось. Теперь у некоторых детей помимо отца могут быть две матери: суррогатная и «юридическая». А если для зачатия используются яйцеклетка и сперматозоид пары, которые затем переносятся в суррогатную мать, у ребенка тоже две матери — генетическая и суррогатная. Теоретически возможны ситуации, когда юридическая, генетическая и суррогатная мать — это три разных человека.
Теоретически возможны ситуации, когда юридическая, генетическая и суррогатная мать — это три разных человека.
Юридические нюансы, возникающие в подобных случаях, уже получают оценки. Например, «Baby M Case»: у Элизабет Стерн был рассеянный склероз, который несет много рисков при беременности, поэтому семья Стерн обратилась в один из медицинских центров в Нью-Йорке для суррогатного материнства. Использовался генетический материал отца. Между сторонами был подписан договор, что юридическими родителями будут Стерны. Но вскоре после рождения суррогатная мать Мэри Бет Вайтхед под угрозой суицида потребовала вернуть ей ребенка. В дело включились полиция и суд. В итоге суд признал Стернов официальными родителями ребенка, но дал суррогатной матери возможность посещения ребенка. Интересно, что основной мотивацией суда было преследование «лучших интересов ребенка». В другой схожей истории, тоже произошедшей в Нью-Йорке, суд рассудил иначе: двое родителей лучше трех, решил суд, отказав в правах посещения ребенка суррогатной матери. Есть деталь: во втором случае пара использовала собственные яйцеклетку и сперму, которые были перенесены в суррогатную мать.
Есть деталь: во втором случае пара использовала собственные яйцеклетку и сперму, которые были перенесены в суррогатную мать.
«Офшоры» для геномного редактирования
Недавно мир потрясло рождение ребенка от трех генетических родителей: в апреле 2016 года появился на свет ребенок, зачатие которого происходило с использованием митохондриальной ДНК третьего человека. Такая процедура была необходима, так как у матери ребенка есть патогенные мутации в митохондриях (органеллы внутри клеток человека, отвечающие за обеспечение клеток энергией, обладают собственным небольшим геномом, передаются ребенку от матери), которые могли привести к появлению у ребенка синдрома Лея, наследственному заболеванию, связанному с поражением ЦНС и энцефалопатией. Два первых ребенка матери погибли от синдрома Лея. Американский врач Джон Чанг, из клиники в Нью-Йорке вместе с родителями из Иордании прибыли в Мексику. И в Иордании, и в США подобные модификации генетического материала были запрещены.
Получается, в современном мире появляются «биомедицинские офшоры». Люди едут в страны с лояльным законодательством для осуществления процедур, неоднозначных с точки зрения этики и допустимости законодательством той или иной страны. Генная терапия уже становится в центре подобных «спорных» случаев. Например, американка Лиз Пэрриш утверждает, что прошла в Колумбии процедуру по редактированию специальных участков ДНК-теломер при помощи вируса. Длина теломер коррелирует со старением. Пэрриш стала первым человеком, решившимся на генетическую терапию для борьбы со старением, до нее эксперименты проводились только на животных. Научное сообщество отнеслось к самовольным клиническим испытаниям Пэрриш неоднозначно, многие подвергли ее действия критике.
В целом на данный момент мировое сообщество с очень большой осторожностью относится к редактированию генома, когда оно не связано напрямую с лечением тяжелых заболеваний, которые нельзя вылечить иным способом. Дело в том, что технологии еще несовершенны и не максимально специфичны. Так, в уже упомянутом эксперименте китайских ученых над эмбрионами в ДНК многих эмбрионов изменились не только участки, которые планировали изменить ученые, но и другие, случайные. Или, например, когда во Франции генную терапию решили применить для лечения врожденного X-сцепленного иммунодефицита, в ходе клинического испытания неожиданно в качестве побочного эффекта у больного развилась лейкемия.
Дело в том, что технологии еще несовершенны и не максимально специфичны. Так, в уже упомянутом эксперименте китайских ученых над эмбрионами в ДНК многих эмбрионов изменились не только участки, которые планировали изменить ученые, но и другие, случайные. Или, например, когда во Франции генную терапию решили применить для лечения врожденного X-сцепленного иммунодефицита, в ходе клинического испытания неожиданно в качестве побочного эффекта у больного развилась лейкемия.
Ребенок на заказ
В целом медицинское и научное сообщество сейчас более лояльно к генной терапии, которая будет влиять только на генетический материал самого человека. Генные модификации, которые передавались бы детям, все еще изучены недостаточно и остаются в «серой зоне». Но в определенной степени выбор определенных черт ребенка с помощью генетических технологий доступен уже сейчас.
В ходе искусственного оплодотворения можно пройти процедуру преимплантационной генетической диагностики или преимплантационного генетического скрининга. При ЭКО (искусственном оплодотворении, вне тела матери, с последующим переносом 2-5-дневного эмбриона в полость матки) на сегодняшний день оплодотворяется несколько яйцеклеток. Можно исследовать геном каждой из них и выбрать наиболее «подходящие» эмбрионы. Такая процедура уже довольно активно используется для профилактики тяжелых наследственных патологий у семей с соответствующими рисками. Однако данная технология, очевидно, может быть применена для выбора черт, не связанных со здоровьем — например, цвета глаз или волос. Это, безусловно, несколько пугающая ситуация, заставляет задуматься о способах применения новых генетических технологий для евгеники, для других манипуляций, описанных фантастами в антиутопиях. Разные страны уже вырабатывают позицию по влиянию родителей на генетические данные своих детей.
При ЭКО (искусственном оплодотворении, вне тела матери, с последующим переносом 2-5-дневного эмбриона в полость матки) на сегодняшний день оплодотворяется несколько яйцеклеток. Можно исследовать геном каждой из них и выбрать наиболее «подходящие» эмбрионы. Такая процедура уже довольно активно используется для профилактики тяжелых наследственных патологий у семей с соответствующими рисками. Однако данная технология, очевидно, может быть применена для выбора черт, не связанных со здоровьем — например, цвета глаз или волос. Это, безусловно, несколько пугающая ситуация, заставляет задуматься о способах применения новых генетических технологий для евгеники, для других манипуляций, описанных фантастами в антиутопиях. Разные страны уже вырабатывают позицию по влиянию родителей на генетические данные своих детей.
В Китае, например, запрещено использование преимплантационной генетической диагностики для выбора пола будущего ребенка. Но такая процедура не запрещена в Штатах. Но волнуют китайцев этические вопросы или для подобного законодательного регулирования важнее демографические причины — большой вопрос.
Но волнуют китайцев этические вопросы или для подобного законодательного регулирования важнее демографические причины — большой вопрос.
Но важно, чтобы свои взгляды на границы генетических вмешательств сформировали не только правительства, но и обычные люди. Иначе мы рискуем оказаться в ситуации неосведомленности населения о базовых принципах генетических технологий и распространения предрассудков. Недавняя волна заявлений об опасности ГМО для человека — яркое тому подтверждение. Один из опросов в Казани, например, показал, что почти половина респондентов считают, что «любые пищевые продукты, содержащие гены», должны быть изъяты из продажи и не должны импортироваться или производиться в стране. Очевидно, что гены есть в любом живом организме, так что такие результаты исследования просто плачевны. Впрочем, 15% опрошенных честно признались, что не представляют себе, что такое ГМО. Ученым, биомедикам и просто тем, кто верит в то, что технологии делают нашу жизнь лучше, теперь нужно сделать все, чтобы генетическое редактирование человека не столкнулось с той же волной необоснованной паники, а стало действительно эффективным инструментом в борьбе с болезнями.
Просто о сложном: CRISPR/Cas
Инфографика на конкурс «био/мол/текст»: CRISPR/Cas — система адаптивного иммунитета бактерий и архей, которая пригодилась и эукариотам. Мы попытались предельно ясно отразить этот механизм, породивший взрыв в биологическом сообществе и, вероятно, сильно изменивший будущее науки и человечества. Из этой инфографики вы узнаете краткую историю изучения, механизм и возможные применения системы CRISPR/Cas.
Эта работа заслужила приз зрительских симпатий конкурса «био/мол/текст»-2016.
В конкурсе участвовала только инфографика!
Текст написала Ольга Волкова.
Генеральным спонсором конкурса, согласно нашему краудфандингу, стал предприниматель Константин Синюшин, за что ему огромный человеческий респект!
Спонсором приза зрительских симпатий выступила фирма «Атлас».
Спонсор публикации этой статьи — Дмитрий Геннадиевич Калашников.
Как устроена иммунная система прокариот?
Системы CRISPR-Cas обнаружены почти у всех известных архей и половины бактерий. Чаще они находятся на хромосоме, реже — в составе фагов (вирусов бактерий) и других мобильных генетических элементов. Эти системы состоят из двух основных блоков: CRISPR-кассеты и прилегающего к ней кластера генов cas. Кассета — это блок прямых почти палиндромных («зеркальных», взаимокомплементарных последовательностей, способных складываться в шпильки) повторов размером 24–48 пар нуклеотидов. Эти повторы перемежаются спейсерами — уникальными вставками примерно такой же длины. Спейсеры идентичны различным участкам фагов и других мобильных элементов, когда-либо проникавших в эту клетку или ее предков. Число повторов в разных системах варьирует от единиц до сотен.
Чаще они находятся на хромосоме, реже — в составе фагов (вирусов бактерий) и других мобильных генетических элементов. Эти системы состоят из двух основных блоков: CRISPR-кассеты и прилегающего к ней кластера генов cas. Кассета — это блок прямых почти палиндромных («зеркальных», взаимокомплементарных последовательностей, способных складываться в шпильки) повторов размером 24–48 пар нуклеотидов. Эти повторы перемежаются спейсерами — уникальными вставками примерно такой же длины. Спейсеры идентичны различным участкам фагов и других мобильных элементов, когда-либо проникавших в эту клетку или ее предков. Число повторов в разных системах варьирует от единиц до сотен.
Таким образом, CRISPR можно считать коллекцией разделенных повторами «фотографий» нарушителей клеточных границ. Составляется эта коллекция простым заимствованием их кусочков, а чтобы противостоять новой инвазии этих же молекулярных агентов, коллекция должна регулярно «просматриваться» и обновляться. Для этой функции нужна лидерная последовательность, предшествующая череде повторов. Она богата «легкоплавкими» АТ-пáрами и содержит промотор, контролирующий транскрипцию CRISPR-кассеты («просмотр коллекции»).
Для этой функции нужна лидерная последовательность, предшествующая череде повторов. Она богата «легкоплавкими» АТ-пáрами и содержит промотор, контролирующий транскрипцию CRISPR-кассеты («просмотр коллекции»).
Гены cas кодируют белки, берущие на себя всю тяжесть работы по встраиванию спейсеров и уничтожению агентов с идентичными последовательностями (протоспейсерами) и помогающие процессировать CRISPR-транскрипт: разделять фото-гирлянду на отдельные портреты. Функцию уничтожения выполняют Cas-белки, называемые эффекторными. В зависимости от типа эффекторов все CRISPR-системы разделяют на два класса: у I класса мишень уничтожается мультибелковым комплексом, а у II — одним крупным белком. Далее эти классы подразделяются на шесть типов. Большинство эффекторов атакует ДНК, лишь один — исключительно РНК [10], редкие — обе молекулы. Один организм может содержать несколько разных систем, а спейсеры различаются в разных клетках даже одной популяции .
К чему это приводит, можно узнать из конкурсной статьи о бактериофагах и вечной гонке вооружений в фаговом и бактериальном мирах: «Пожиратели бактерий: убийцы в роли спасителей» [11]. Кстати, там много интересных авторских электронных изображений фагов.
Для решения инженерных задач больше всего подходит система II типа, относящаяся ко II классу, — она самая простая. Именно ее эффекторный белок называется Cas9 — то самое обозначение, что фигурирует в современных системах редактирования генома.
Как формируется CRISPR-опосредованный иммунитет?
Если в бактерию или архею, снабженную CRISPR-системой, проникает вирус, включается адаптационный функциональный модуль системы: специфические Cas-белки — у всех систем это как минимум Cas1 и Cas2 — вырезают из чужака понравившиеся фрагменты. Подобрать протоспейсер в некоторых случаях помогает и эффекторный белок. Белки выбирают участки рядом с особой последовательностью PAM (protospacer adjacent motif) — всего несколько нуклеотидов, но неодинаковых для разных CRISPR-систем. Затем эти же адаптационные белки встраивают фрагмент в CRISPR-кассету, всегда с одной стороны — у лидерной последовательности. Так образуется новый спейсер, а заодно с ним — и новый повтор. Весь этот процесс называют адаптацией, или приобретением, а по сути это — запоминание врага. Информацию обо всех запомнившихся врагах получает при делениях всё потомство клетки.
Затем эти же адаптационные белки встраивают фрагмент в CRISPR-кассету, всегда с одной стороны — у лидерной последовательности. Так образуется новый спейсер, а заодно с ним — и новый повтор. Весь этот процесс называют адаптацией, или приобретением, а по сути это — запоминание врага. Информацию обо всех запомнившихся врагах получает при делениях всё потомство клетки.
Как реализуется CRISPR-опосредованный иммунитет?
Для поиска повторно вторгающихся агентов CRISPR-кассета должна экспрессироваться. В результате ее транскрипции образуется длинная молекула РНК — pre-crРНК. С помощью РНКазы III и, как правило, Cas-белков транскрипт нарезается по повторам на отдельные crРНК — молекулы, содержащие один спейсер и кусочки окружающих его повторов (один из них длиннее). В системах II типа для этого процесса, называемого созреванием, необходим еще один участник — tracrРНК (trans-activating CRISPR RNA), которая закодирована рядом с cas-кластером [12].
Далее у систем I класса crРНК взаимодействует с комплексом Cas-белков, а у систем II класса crРНК либо дуплексы tracrРНК-crРНК связываются с одним белком-эффектором, например Cas9. Так образуется интерференционный функциональный модуль — рабочая иммунная единица, состоящая из направляющей РНК и эффекторного белка (или комплекса). Совокупность таких единиц «сканирует» клетку в поисках интервентов.
При обнаружении комплементарной crРНК последовательности, то есть протоспейсера, модуль «слипается» с ней и определяет, не помечена ли она как «своя», клеточная. Если нет, и если к ней прилегает тот самый PAM, то эффекторный белок, который представляет собой эндонуклеазу, разрезает обе цепи ДНК в строго определенных местах. Весь процесс называется интерференцией. В особом случае, у системы VI типа, происходит РНК-интерференция, потому что эффекторный белок является рибонуклеазой и разрушает РНК. Так или иначе, атакованные фаги или плазмиды выводятся из строя. Ну и появляется лишняя возможность «наворовать» новые спейсеры.
Ну и появляется лишняя возможность «наворовать» новые спейсеры.
Какие проблемы могут возникнуть при реализации иммунного ответа? Не исключено, что по мере удаления от лидерной последовательности, то есть от CRISPR-промотора, шансы спейсера транскрибироваться и созреть уменьшаются. Кроме того, есть мнение, что удаленные спейсеры со временем могут накапливать мутации, препятствующие эффективной интерференции с мишенью, или вовсе удаляться. Но раз адаптация новых спейсеров происходит вблизи промотора, удаленные спейсеры представляют собой фото агентов, давно не нападавших на эту клеточную линию, и в постоянной боеготовности по отношению к ним клетка не нуждается. Настоящей же проблемой могут стать даже однонуклеотидные мутации мишени. В общем, комплементарность в этом деле превыше всего.
А не приручить ли нам чужой иммунитет?
Детально изучив принципы работы стрептококковой системы CRISPR-Cas9 (II тип), ученые подумали: а почему бы не попробовать с ее помощью корректировать геномы других организмов? Появились новые надежды относительно лечения генетических (и не только) заболеваний человека, ведь этот способ редактирования in vivo мог оказаться эффективнее уже вовсю тестируемых в то время нуклеаз ZFN и TALEN [13].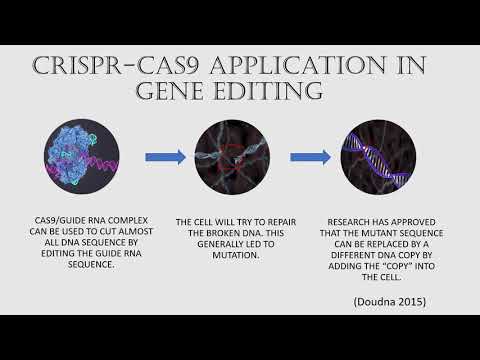
Всё, что требовалось для новой технологии, — это разместить на векторах ген белка Cas9 и CRISPR-кассету, где спейсеры сделать идентичными местам генома, которые нужно изменить. Меняя число и тип спейсеров, можно модифицировать сразу несколько разных участков генома. Довольно быстро поняли, что tracrРНК и crРНК можно безболезненно объединить в одну химерную молекулу sgРНК (single-guide RNA), а РНКазу III в эукариотических клетках спокойно подменяют другие рибонуклеазы. Ну и еще потребовалось оптимизировать систему для эукариотических клеток: подправить кодонный состав и добавить ядерный «адрес», чтобы она четко следовала к месту работы — хромосомам.
Получилась простая и, что немаловажно, дешевая двухкомпонентная система: ген cas9 и CRISPR-кассета транскрибируются в клеточном ядре выбранного организма, CRISPR-транскрипт нарезается на отдельные sgРНК, которые объединяются с белками Cas9 и ищут цель. Когда sgРНК находит комплементарный участок в геноме организма, Cas9 разрезает «натупо» обе цепи ДНК. Всё, работа CRISPR-системы на этом окончена. Теперь эстафета передается репарационным системам самогό организма. Они решают, как лучше залатать разрез: то ли просто сшить куски (это будет негомологичное соединение концов, NHEJ), то ли, если есть подходящая матрица с флангами, комплементарными участкам ДНК с двух сторон от разрыва, поставить «заплатку» (это будет гомологичная рекомбинация). Так вот, первый вариант выгоден, если нужно что-то вырезать, второй — если нужно что-то вставить или заменить дефектный участок ДНК на нормальный, который просто вводят на подходящем векторе. Иногда используют гомологию с парной хромосомой, если на ней нужный локус не дефектный.
Всё, работа CRISPR-системы на этом окончена. Теперь эстафета передается репарационным системам самогό организма. Они решают, как лучше залатать разрез: то ли просто сшить куски (это будет негомологичное соединение концов, NHEJ), то ли, если есть подходящая матрица с флангами, комплементарными участкам ДНК с двух сторон от разрыва, поставить «заплатку» (это будет гомологичная рекомбинация). Так вот, первый вариант выгоден, если нужно что-то вырезать, второй — если нужно что-то вставить или заменить дефектный участок ДНК на нормальный, который просто вводят на подходящем векторе. Иногда используют гомологию с парной хромосомой, если на ней нужный локус не дефектный.
Разумеется, технология пока не лишена недостатков. Cas9, например, может проявлять нецелевую активность, «закрывая глаза» на мелкие несоответствия между sgРНК и мишенью. По словам К. Северинова, основная проблема — это биоинформатическое предсказание мишеней, поскольку, помимо наличия участка PAM, необходимо учитывать массу факторов, включая состояние хроматина. Кроме того, сценарий, по которому пойдет репарация разреза, не всегда соответствует желаемому, поэтому сейчас активно ищут факторы, влияющие на выбор этого сценария клеткой. Помимо оптимизации CRISPR-Cas9 и механизмов ее доставки в нужные клетки, ведется апробирование других типов CRISPR-систем [14].
Кроме того, сценарий, по которому пойдет репарация разреза, не всегда соответствует желаемому, поэтому сейчас активно ищут факторы, влияющие на выбор этого сценария клеткой. Помимо оптимизации CRISPR-Cas9 и механизмов ее доставки в нужные клетки, ведется апробирование других типов CRISPR-систем [14].
Спектр применений CRISPR-Cas9 и ее модификаций
Точки приложения CRISPR-технологии можно условно объединить в три крупные группы: «CRISPR — для исследований», «CRISPR — для биотехнологий» и «CRISPR — для терапии».
1. «CRISPR — для исследований». Технология позволяет изучать роль конкретных генов в процессах развития и жизнедеятельности организмов. Как вариант — устанавливать роль генов и их перестроек в возникновении и прогрессировании генетических болезней и рака: этот инструмент позволяет создавать прекрасные модельные системы [15].
Если Cas9 лишают одного нуклеазного домена, то белок становится никазой (nCas9) — режет только одну цепь ДНК, — а если лишают сразу двух, то белок становится инактивированным, или «мертвым» (dead, dCas9). Такой белок ничего не режет, зато систему CRISPR-dCas9 можно использовать для репрессии целых наборов генов или как платформу для конструирования более сложных регуляторных и модифицирующих комплексов. Например, если к ней привязать активирующий домен, то экспрессия целевых генов активируется. Для эпигенетической модификации нужных зон достаточно добавить модифицирующий домен. А пометив dCas9 флюоресцентными белками [16], можно визуализировать разные области хромосом. Ясно, что регуляторные возможности системы будут востребованы и в медицине. Кроме того, разные варианты CRISPR-Cas открывают новые возможности для скрининга мишеней лекарств [1].
Такой белок ничего не режет, зато систему CRISPR-dCas9 можно использовать для репрессии целых наборов генов или как платформу для конструирования более сложных регуляторных и модифицирующих комплексов. Например, если к ней привязать активирующий домен, то экспрессия целевых генов активируется. Для эпигенетической модификации нужных зон достаточно добавить модифицирующий домен. А пометив dCas9 флюоресцентными белками [16], можно визуализировать разные области хромосом. Ясно, что регуляторные возможности системы будут востребованы и в медицине. Кроме того, разные варианты CRISPR-Cas открывают новые возможности для скрининга мишеней лекарств [1].
2. «CRISPR — для биотехнологий». Здесь речь идет о применении CRISPR-Cas9 как минимум для трех целей:
- для улучшения свойств сельскохозяйственных животных и растений. Уже создали и протестировали CRISPR-системы для риса, пшеницы, кукурузы, сорго и многих других культур. Детально эта кухня разобрана в обзоре [17].
 Помимо улучшения пищевых качеств, такими инструментами легко наделять культуры устойчивостью к вредителям и химикалиям, а животных — избавлять от нежелательных генов. Например, недавно в свиных клетках инактивировали эндогенных ретровирусов (не то чтобы здоровья свиньи ради, а держа в голове планы по трансплантации ее органов человеку) [18];
Помимо улучшения пищевых качеств, такими инструментами легко наделять культуры устойчивостью к вредителям и химикалиям, а животных — избавлять от нежелательных генов. Например, недавно в свиных клетках инактивировали эндогенных ретровирусов (не то чтобы здоровья свиньи ради, а держа в голове планы по трансплантации ее органов человеку) [18]; - для контроля распространения инфекций, переносимых животными. Например, уже всерьез размышляют о «заносе» в природные популяции малярийных комаров генов устойчивости к плазмодию или генов популяционного контроля [19]. Это стало принципиально возможным благодаря технологии «gene drive» (что-то типа «продвижения гена»), основанной на изменении классического наследования. С ее помощью встроенный в одну особь ген быстро распространяется по всей популяции. Принцип технологии иллюстрирует нашумевшая мутагенная цепная реакция у дрозофил [20];
- для конструирования новых метаболических путей и осуществления направленной эволюции биомолекул.
 Новые или оптимизированные ферментные системы бактерий и грибов, получаемые так легко и дешево, — предел мечтаний технологов из целого ряда отраслей промышленности. Но даже простое встраивание в промышленно важные штаммы бактерий систем CRISPR-Cas с заданными свойствами может защитить их от бактериофагов и нежелательных плазмид.
Новые или оптимизированные ферментные системы бактерий и грибов, получаемые так легко и дешево, — предел мечтаний технологов из целого ряда отраслей промышленности. Но даже простое встраивание в промышленно важные штаммы бактерий систем CRISPR-Cas с заданными свойствами может защитить их от бактериофагов и нежелательных плазмид.
3. «CRISPR — для терапии». Здесь пределов для фантазии, кажется, и вовсе нет. Если говорить о наследственных заболеваниях, то CRISPR-Cas9 в культурах клеток или животных моделях уже «примерили» для серповидноклеточной анемии и β-талассемии, M2DS-синдрома и миодистрофии Дюшенна, муковисцидоза (исправили мутантный CFTR-локус в кишечных стволовых клетках человека) и тирозинемии, катаракты (у мышей устранили доминантную мутацию в гене Crygc) и пигментного ретинита. Вообще, болезни глаз сейчас в центре внимания, потому что в глаза генетические конструкции легко доставлять [14].
Преимущества коррекции генома в зародышевой линии (как совокупности любых генеративных клеток, связывающих друг с другом поколения организмов) и стволовых клетках очевидны, но даже изменения, вносимые в соматические клетки уже развитых органов, дают эффект. Особенно если речь идет о борьбе с болезнями печени и мышц. О результатах терапевтического применения CRISPR-Cas9 в разных типах клеток рассказывает свежий обзор [21].
Особенно если речь идет о борьбе с болезнями печени и мышц. О результатах терапевтического применения CRISPR-Cas9 в разных типах клеток рассказывает свежий обзор [21].
Отдельное перспективное направление — борьба с хроническими вирусными заболеваниями типа гепатитов и ВИЧ-инфекции. Если возбудитель сохраняется в организме в виде провируса (вирусной ДНК, встроенной в клеточный геном), то его можно просто вырезать. Именно так и поступил коллектив биологов из США, избавив лимфоциты человека от ВИЧ (об этом сообщили сразу две «биомолекулярные» статьи: «Битва века: CRISPR VS ВИЧ» [22] и «CRISPR/Cas9 как помощник в борьбе с ВИЧ» [23]). Правда, ВИЧ — объект крайне изменчивый, и с ним еще придется поломать копья.
Можно помечтать, что в терапии опухолей найдут применение варианты недавно описанной CRISPR-системы VI типа — той, что уничтожает только РНК, причем, как оказалось, любую клеточную РНК без разбора: запустить такую систему в раковую клетку — это как наслать на нее проклятье [14].
CRISPR-Cas — это не только иммунитет
Оказывается, для бактерий и их эволюции эта система значит намного больше.
Неканонические активности CRISPR-систем или их отдельных компонентов возникали как побочные продукты их иммунной функции либо как самостоятельно отбираемые признаки. Скорее всего, CRISPR-кассеты и Cas-белки когда-то работали порознь, причем исходная задача последних состояла в регуляции экспрессии генов и репарации ДНК [7]. Современные компоненты CRISPR-Cas замечены:
- в регуляции активности генов. Эти системы могут вмешиваться в общение бактерий по типу quorum sensing («чувство кворума») [24] и регулировать таким образом групповое поведение: формирование плодовых тел и спор у миксококков и биопленок у синегнойной палочки. Белки Cas9 (систем II типа) регулируют вирулентность патогенов Legionella pneumophila, Francisella novicida, Campylobacter jejuni и, возможно, Neisseria meningitidis;
- в репарации ДНК.
 Cпособность к разрезанию CRISPR-кассеты для встраивания новых спейсеров, скорее всего, вторичное функциональное приобретение Cas1. Исходно он резал типичные рекомбинационные/репарационные интермедиаты и пока что не разучился это делать. Поэтому экспрессия генов cas повышает устойчивость некоторых бактерий к радиации, а отключение CRISPR-системы ведет к повышению их чувствительности к повреждающим ДНК факторам и нарушению расхождения хромосом;
Cпособность к разрезанию CRISPR-кассеты для встраивания новых спейсеров, скорее всего, вторичное функциональное приобретение Cas1. Исходно он резал типичные рекомбинационные/репарационные интермедиаты и пока что не разучился это делать. Поэтому экспрессия генов cas повышает устойчивость некоторых бактерий к радиации, а отключение CRISPR-системы ведет к повышению их чувствительности к повреждающим ДНК факторам и нарушению расхождения хромосом; - в ремоделировании (реорганизации) генома. Cas-белки иногда ошибаются и вместо вражеской ДНК делают спейсерами фрагменты своего генома. Если последовавшие за этим «аутоиммунные» реакции не приводят к гибели клетки, то чаще всего происходят крупные перестройки, направленные на частичное или полное избавление от «проштрафившейся» CRISPR-системы. Иногда перестройки могут повысить приспособленность хозяина к нише — например, за счет дупликации полезных генов;
- в конкуренции мобильных генетических элементов друг с другом, если они несут эти самые CRISPR-Cas-системы;
- во введении бактерий в «спящее», неактивное состояние.
 Функция эта по сути тоже иммунная, но экстремальная. Предполагают, что в избранных CRISPR-системах один из Cas-белков может служить «токсином», а его партнер — «антитоксином», и когда в клетку проникает фаг, «токсин» высвобождается и начинает крушить любую РНК. Но у «замирающей» при этом клетки есть время лихорадочно насобирать спейсеры. Если это не получается, то разбушевавшийся Cas-токсин доводит клетку до самоубийства. Доказать этот принцип пока не удалось [7]. Зато подобный драматический финал мы уже где-то видели: именно так поступает система VI типа. Такой исход фаговой инвазии называется абортивной инфекцией. Для клетки плохо, для популяции — хорошо…
Функция эта по сути тоже иммунная, но экстремальная. Предполагают, что в избранных CRISPR-системах один из Cas-белков может служить «токсином», а его партнер — «антитоксином», и когда в клетку проникает фаг, «токсин» высвобождается и начинает крушить любую РНК. Но у «замирающей» при этом клетки есть время лихорадочно насобирать спейсеры. Если это не получается, то разбушевавшийся Cas-токсин доводит клетку до самоубийства. Доказать этот принцип пока не удалось [7]. Зато подобный драматический финал мы уже где-то видели: именно так поступает система VI типа. Такой исход фаговой инвазии называется абортивной инфекцией. Для клетки плохо, для популяции — хорошо…
- J. A. Doudna, E. Charpentier. (2014). The new frontier of genome engineering with CRISPR-Cas9. Science. 346, 1258096-1258096;
- Ruud. Jansen, Jan. D. A. van Embden, Wim. Gaastra, Leo. M. Schouls. (2002). Identification of genes that are associated with DNA repeats in prokaryotes.
 Mol Microbiol. 43, 1565-1575;
Mol Microbiol. 43, 1565-1575; - Puping Liang, Yanwen Xu, Xiya Zhang, Chenhui Ding, Rui Huang, et. al.. (2015). CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 6, 363-372;
- Erik J. Sontheimer, Rodolphe Barrangou. (2015). The Bacterial Origins of the CRISPR Genome-Editing Revolution. Human Gene Therapy. 26, 413-424;
- Jennifer L. Gori, Patrick D. Hsu, Morgan L. Maeder, Shen Shen, G. Grant Welstead, David Bumcrot. (2015). Delivery and Specificity of CRISPR/Cas9 Genome Editing Technologies for Human Gene Therapy. Human Gene Therapy. 26, 443-451;
- CRISPR-системы: иммунизация прокариот;
- Edze R. Westra, Angus Buckling, Peter C. Fineran. (2014). CRISPR–Cas systems: beyond adaptive immunity. Nat Rev Micro. 12, 317-326;
- Eric S.
 Lander. (2016). The Heroes of CRISPR. Cell. 164, 18-28;
Lander. (2016). The Heroes of CRISPR. Cell. 164, 18-28; - Rongxue Peng, Guigao Lin, Jinming Li. (2016). Potential pitfalls of CRISPR/Cas9-mediated genome editing. FEBS J. 283, 1218-1231;
- Omar O. Abudayyeh, Jonathan S. Gootenberg, Silvana Konermann, Julia Joung, Ian M. Slaymaker, et. al.. (2016). C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353, aaf5573;
- Пожиратели бактерий: убийцы в роли спасителей;
- Элементы: «Прокариотическая система иммунитета поможет редактировать геном»;
- А не замахнуться ли нам на… изменение генома?;
- Ершов А. (2016). «Просто это очень красиво». Константин Северинов о новом типе CRISPR-систем и последних трендах в редактировании геномов. Сайт N+1;
- Andrea Pellagatti, Hamid Dolatshad, Simona Valletta, Jacqueline Boultwood.
 (2015). Application of CRISPR/Cas9 genome editing to the study and treatment of disease. Arch Toxicol. 89, 1023-1034;
(2015). Application of CRISPR/Cas9 genome editing to the study and treatment of disease. Arch Toxicol. 89, 1023-1034; - Флуоресцентные белки: разнообразнее, чем вы думали!;
- Xingliang Ma, Qinlong Zhu, Yuanling Chen, Yao-Guang Liu. (2016). CRISPR/Cas9 Platforms for Genome Editing in Plants: Developments and Applications. Molecular Plant. 9, 961-974;
- L. Yang, M. Guell, D. Niu, H. George, E. Lesha, et. al.. (2015). Genome-wide inactivation of porcine endogenous retroviruses (PERVs). Science. 350, 1101-1104;
- Kyle Jarrod McLean, Marcelo Jacobs-Lorena. (2016). Genetic Control Of Malaria Mosquitoes. Trends in Parasitology. 32, 174-176;
- Мутагенная цепная реакция: редактирование геномов на грани фантастики;
- Nataša Savić, Gerald Schwank. (2016). Advances in therapeutic CRISPR/Cas9 genome editing.
 Translational Research. 168, 15-21;
Translational Research. 168, 15-21; - Битва века: CRISPR vs ВИЧ;
- CRISPR/Cas9 как помощник в борьбе с ВИЧ;
- Бактерии, молчать! Как и зачем вносить помехи в межклеточное общение.
Система CRISPR/Cas9 – универсальный инструмент геномной инженерии | Смирнов
1. Alphey L. Can CRISPR-Cas9 gene drives curb malaria? Nat. Biotechnol. 2016;34(2):149-150.
2. Anders C., Niewoehner O., Duerst A., Jinek M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 2014;513(7519):569-573.
3. Bakondi B., Lv W., Lu B., Jones M.K., Tsai Y., Kim K.J., Levy R., Akhtar A.A., Breunig J.J., Svendsen C.N., Wang S. In vivo CRISPR/Cas9 gene editing corrects retinal dystrophy in the S334ter-3 rat model of autosomal dominant retinitis pigmentosa. Mol. Ther.: J. Am. Soc. Gene Ther. 2015;24(3):556-563.
4. Barrangou R., Fremaux C., Deveau H. , Richards M., Boyaval P., Moineau S., Romero D.A., Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science (N.Y.). 2007; 315(5819):1709-1712.
, Richards M., Boyaval P., Moineau S., Romero D.A., Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science (N.Y.). 2007; 315(5819):1709-1712.
5. Bassett A.R., Tibbit C., Ponting C.P., Liu J.L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 2013;4(1):220-228.
6. Bassuk A.G., Zheng A., Li Y., Tsang S.H., Mahajan V.B. Precision medicine: genetic repair of retinitis pigmentosa in patient-derived stem cells. Sci. Rep. 2016;6:19969.
7. Bauer D.E., Canver M.C., Orkin S.H. Generation of genomic deletions in mammalian cell lines via CRISPR/Cas9. J. Vis. Exp. 2014;83:e52118.
8. Bolukbasi M.F., Gupta A., Wolfe S.A. Creating and evaluating accurate CRISPR-Cas9 scalpels for genomic surgery. Nat. Methods. 2016; 13(1):37-40.
9. Boutros M., Ahringer J. The art and design of genetic screens: RNA interference. Nat. Rev. Genet. 2008;9(7):554-566.
10. Brandl C., Ortiz O., Rottig B., Wefers B., Wurst W. , Kuhn R. Creation of targeted genomic deletions using TALEN or CRISPR/Cas nuclease pairs in one-cell mouse embryos. FEBS Open Bio. 2015;5:26-35.
, Kuhn R. Creation of targeted genomic deletions using TALEN or CRISPR/Cas nuclease pairs in one-cell mouse embryos. FEBS Open Bio. 2015;5:26-35.
11. Burt A. Site-specific selfish genes as tools for the control and genetic engineering of natural populations. Proc. Roy. Soc. Lond. 2003;270(1518):921-928.
12. Canver M.C., Bauer D.E., Dass A., Yien Y.Y., Chung J., Masuda T., Maeda T., Paw B.H., Orkin S.H. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/cas9 nuclease system in mammalian cells. J. Biol. Chem. 2014;289(31):21312-21324.
13. Champer J., Buchman A., Akbari O.S. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nat. Rev. Genet. 2016;17(3):146-159.
14. Chen B., Gilbert L.A., Cimini B.A., Schnitzbauer J., Zhang W., Li G.W., Park J., Blackburn E.H., Weissman J.S., Qi L.S., Huang B. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 2013;155(7):1479-1491.
Cell. 2013;155(7):1479-1491.
15. Chen S., Sanjana N.E., Zheng K., Shalem O., Lee K., Shi X., Scott D.A., Song J., Pan J.Q., Weissleder R., Lee H., Zhang F., Sharp P.A. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell. 2015;160(6):1246-1260.
16. Cheng A.W., Wang H., Yang H., Shi L., Katz Y., Theunissen T.W., Rangarajan S., Shivalila C.S., Dadon D.B., Jaenisch R. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 2013;23(10):1163-1171.
17. Cho S.W., Kim S., Kim Y., Kweon J., Kim H.S., Bae S., Kim J.-S. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 2014;24(1):132-141.
18. Choi P.S., Meyerson M. Targeted genomic rearrangements using CRISPR/Cas technology. Nat. Commun. 2014;5:3728.
19. Chu V.T., Weber T., Wefers B., Wurst W., Sander S., Rajewsky K., Kühn R. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat. Biotechnol. 2015;33(5):543-548.
Nat. Biotechnol. 2015;33(5):543-548.
20. Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W., Marraffini L.A., Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013;339(6121): 819-823.
21. Costello M.J., May R.M., Stork N.E. Can we name Earth’s species before they go extinct? Science. 2013;339(6118):413-416.
22. Cox D.B.T., Platt R.J., Zhang F. Therapeutic genome editing: prospects and challenges. Nat. Med. 2015;21(2):121-131.
23. Crispo M., Mulet A.P., Tesson L., Barrera N., Cuadro F., dos SantosNeto P.C., Nguyen T.H., Crénéguy A., Brusselle L., Anegón I., Menchaca A. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes. PloS ONE. 2015;10(8):e0136690.
24. Cyranoski D. Embryo editing divides scientists. Nature. 2015;519 (7543):272.
25. Davis K.M., Pattanayak V., Thompson D.B., Zuris J.A., Liu D.R. Small molecule-triggered Cas9 protein with improved genome-editing specificity. Nat. Chem. Biol. 2015;11(5):316-318.
Nat. Chem. Biol. 2015;11(5):316-318.
26. Deredec A., Burt A., Godfray H.C.J. The population genetics of using homing endonuclease genes in vector and pest management. Genetics. 2008;179(4):2013-2026.
27. Dianov G.L., Hübscher U. Mammalian base excision repair: the forgotten archangel. Nucl. Acids Res. 2013;41(6):3483-3490.
28. Ding Q., Strong A., Patel K.M., Ng S.L., Gosis B.S., Regan S.N., Cowan C.A., Rader D.J., Musunuru K. Permanent alteration of PCSK9 with in vivo CRISPR-Cas9 genome editing. Circ. Res. 2014;115(5):488-492.
29. Doench J.G., Fusi N., Sullender M., Hegde M., Vaimberg E.W., Donovan K.F., Smith I., Tothova Z., Wilen C., Orchard R., Virgin H.W., Listgarten J., Root D.E. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 2016;34(2):184-191.
30. Elliott B., Richardson C., Winderbaum J., Nickoloff J.A., Jasin M. Gene conversion tracts from double-strand break repair in mammalian cells. Mol. Cell. Biol. 1998;18(1):93-101.
Cell. Biol. 1998;18(1):93-101.
31. Essletzbichler P., Konopka T., Santoro F., Chen D., Gapp B.V., Kralovics R., Brummelkamp T.R., Nijman S.M.B., Bürckstümmer T. Megabase-scale deletion using CRISPR/Cas9 to generate a fully haploid human cell line. Genome Res. 2014;24(12):2059-2065.
32. Fei J.F., Schuez M., Tazaki A., Taniguchi Y., Roensch K., Tanaka E.M. CRISPR-mediated genomic deletion of Sox2 in the axolotl shows a requirement in spinal cord neural stem cell amplification during tail regeneration. Stem Cell Reports. 2014;3(3):444-459.
33. Findlay G.M., Boyle E.A., Hause R.J., Klein J.C., Shendure J. Saturation editing of genomic regions by multiplex homology-directed repair. Nature. 2014;513(7516):120-123.
34. Flowers J.J., He S., Malfatti S., del Rio T.G., Tringe S.G., Hugenholtz P., McMahon K.D. Comparative genomics of two “Candidatus Accumulibacter” clades performing biological phosphorus removal. ISME J. 2013;7(12):2301-2314.
35. Frock R.L., Hu J., Meyers R. M., Ho Y.-J., Kii E., Alt F.W. Genomewide detection of DNA double-stranded breaks induced by engineered nucleases. Nat. Biotechnol. 2015;33(2):179-186.
M., Ho Y.-J., Kii E., Alt F.W. Genomewide detection of DNA double-stranded breaks induced by engineered nucleases. Nat. Biotechnol. 2015;33(2):179-186.
36. Fu Y., Foden J.A., Khayter C., Maeder M.L., Reyon D., Joung J.K., Sander J.D. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 2013;31(9): 822-826.
37. Fu Y., Sander J.D., Reyon D., Cascio V.M., Joung J.K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat. Biotechnol. 2014;32(3):279-284.
38. Fujihara Y., Ikawa M. CRISPR/Cas9-based genome editing in mice by single plasmid injection. Method. Enzymol. 2014;546:319-336.
39. Fujii W., Kawasaki K., Sugiura K., Naito K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucl. Acids Res. 2013;41(20):e187.
40. Gantz V.M., Bier E. The mutagenic chain reaction: A method for converting heterozygous to homozygous mutations. Science. 2015; 348(6233):442-444.
41. Gantz V.M., Jasinskiene N., Tatarenkova O., Fazekas A., Macias V.M., Bier E., James A.A. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proc. Natl Acad. Sci. 2015;112(49):E6736-E6743.
42. Geisinger J.M., Turan S., Hernandez S., Spector L.P., Calos M.P. In vivo blunt-end cloning through CRISPR/Cas9-facilitated non-homologous end-joining. Nucl. Acids Res. 2016;44(8):e76.
43. Gonzales A.P.W., Yeh J.R. Cas9-based genome editing in Zebrafish. Methods Enzymol. 2014;546:377-413.
44. González F., Zhu Z., Shi Z.-D., Lelli K., Verma N., Li Q.V, Huangfu D. An iCRISPR platform for rapid, multiplexable, and inducible genome editing in human pluripotent stem cells. Cell Stem Cell. 2014;15(2):215-226.
45. Graham D.B., Root D.E. Resources for the design of CRISPR gene editing experiments. Genome Biol. 2015;16(1):260.
46. Gratz S.J., Wildonger J., Harrison M.M., O’Connor-Giles K.M. CRISPR/Cas9-mediated genome engineering and the promise of designer flies on demand. Fly. 2013;7(4):37-41.
Fly. 2013;7(4):37-41.
47. Guilinger J.P., Thompson D.B., Liu D.R. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat. Biotechnol. 2014;32(6):577-582.
48. Gupta A., Hall V.L., Kok F.O., Shin M., McNulty J.C., Lawson N.D., Wolfe S.A. Targeted chromosomal deletions and inversions in zebrafish. Genome Res. 2013;23(6):1008-1017.
49. Hammond A., Galizi R., Kyrou K., Simoni A., Siniscalchi C., Katsanos D., Gribble M., Baker D., Marois E., Russell S., Burt A., Windbichler N., Crisanti A., Nolan T. A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. Nat. Biotechnol. 2015;34(1):1-8.
50. Han J., Zhang J., Chen L., Shen B., Zhou J., Hu B., Du Y., Tate P.H., Huang X., Zhang W. Efficient in vivo deletion of a large imprinted lncRNA by CRISPR/Cas9. RNA Biology. 2014;11(7):829-835.
51. He Z., Proudfoot C., Mileham A.J., Mclaren D.G., Whitelaw C.B.A., Lillico S. G. Highly efficient targeted chromosome deletions using CRISPR/Cas9. Biotechnol. Bioeng. 2015;112(5):1060-1064.
G. Highly efficient targeted chromosome deletions using CRISPR/Cas9. Biotechnol. Bioeng. 2015;112(5):1060-1064.
52. Hendel A., Bak R.O., Clark J.T., Kennedy A.B., Ryan D.E., Roy S., Steinfeld I., Lunstad B.D., Kaiser R.J., Wilkens A.B., Bacchetta R., Tsalenko A., Dellinger D., Bruhn L., Porteus M.H. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells. Nat. Biotechnol. 2015;33(9):985-989.
53. Hilton I.B., D’Ippolito A.M., Vockley C.M., Thakore P.I., Crawford G.E., Reddy T.E., Gersbach C.A. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat. Biotechnol. 2015;33(5):510-517.
54. Himeda C.L., Jones T.I., Jones P.L. CRISPR/dCas9-mediated transcriptional inhibition ameliorates the epigenetic dysregulation at D4Z4 and represses DUX4-fl in FSH muscular dystrophy. Mol. Ther.: J. Am. Soc. Gene Ther. 2016;24(3):527-535.
55. Hou Z., Zhang Y., Propson N.E., Howden S.E., Chu L.-F., Sontheimer E. J., Thomson J.A. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proc. Natl Acad. Sci. USA. 2013;110(39):15644-15649.
J., Thomson J.A. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proc. Natl Acad. Sci. USA. 2013;110(39):15644-15649.
56. Hsu P.D., Scott D.A., Weinstein J.A., Ran F.A., Konermann S., Agarwala V., Li Y., Fine E.J., Wu X., Shalem O., Cradick T.J., Marraffini L.A., Bao G., Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 2013;31(9):827-832.
57. Hu X., Chang N., Wang X., Zhou F., Zhou X., Zhu X., Xiong J.-W. Heritable gene-targeting with gRNA/Cas9 in rats. Cell Res. 2013; 23(11):1322-1325.
58. Jain I.H., Zazzeron L., Goli R., Alexa K., Schatzman-Bone S., Dhillon H., Goldberger O., Peng J., Shalem O., Sanjana N.E., Zhang F., Goessling W., Zapol W.M., Mootha V.K. Hypoxia as a therapy for mitochondrial disease. Science. 2016;352(6281):54-61.
59. Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J.A., Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012;337(6096):816-821.
Science. 2012;337(6096):816-821.
60. Jinek M., Jiang F., Taylor D.W., Sternberg S.H., Kaya E., Ma E., Anders C., Hauer M., Zhou K., Lin S., Kaplan M., Iavarone A.T., Charpentier E., Nogales E., Doudna J.A. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science. 2014; 343(6176):1247997.
61. Kleinstiver B.P., Pattanayak V., Prew M.S., Tsai S.Q., Nguyen N.T., Zheng Z., Joung J.K. High-fidelity CRISPR–Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 2016;529(7587): 490-495.
62. Konermann S., Brigham M.D., Trevino A.E., Joung J., Abudayyeh O.O., Barcena C., Hsu P.D., Habib N., Gootenberg J.S., Nishimasu H., Nureki O., Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 2014;517(7536):583-588.
63. Kraft K., Geuer S., Will A.J., Chan W., Paliou C., Borschiwer M., Harabula I., Wittler L., Franke M., Ibrahim D.M., Kragesteen B.K., Spielmann M., Mundlos S., Lupianez D.G., Andrey G. Deletions, inversions, duplications: Engineering of structural variants using CRISPR/Cas in mice. Cell Reports. 2015;10(5):833-839.
Deletions, inversions, duplications: Engineering of structural variants using CRISPR/Cas in mice. Cell Reports. 2015;10(5):833-839.
64. Kuscu C., Arslan S., Singh R., Thorpe J., Adli M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat. Biotechnol. 2014;32(7):677-683.
65. Lanphier E., Urnov F.D., Ehlen S.H., Werner M., Smolenski J. Don’t edit the human germ line. Nature. 2015;519:410-411.
66. Li D., Qiu Z., Shao Y., Chen Y., Guan Y., Liu M., Li Y., Gao N., Wang L., Lu X., Zhao Y., Liu M. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nat. Biotechnol. 2013;31(8): 681-683.
67. Liang P., Xu Y., Zhang X., Ding C., Huang R., Zhang Z., Lv J., Xie X., Chen Y., Li Y., Sun Y., Bai Y., Songyang Z., Ma W., Zhou C., Huang J. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 2015;6(5):363-372.
68. Liao H.-K., Gu Y., Diaz A., Marlett J., Takahashi Y., Li M., Suzuki K. , Xu R., Hishida T., Chang C.-J., Esteban C.R., Young J., Izpisua Belmonte J.C. Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells. Nat. Commun. 2015; 6:6413.
, Xu R., Hishida T., Chang C.-J., Esteban C.R., Young J., Izpisua Belmonte J.C. Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells. Nat. Commun. 2015; 6:6413.
69. Lin Y., Cradick T.J., Brown M.T., Deshmukh H., Ranjan P., Sarode N., Wile B.M., Vertino P.M., Stewart F.J., Bao G. CRISPR/ Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucl. Acids Res. 2014;42(11):7473-7485.
70. Liu X., Homma A., Sayadi J., Yang S., Ohashi J., Takumi T. Sequence features associated with the cleavage efficiency of CRISPR/Cas9 system. Sci. Reports. 2016;6:19675.
71. Long C., Amoasii L., Mireault A.A., McAnally J.R., Li H., SanchezOrtiz E., Bhattacharyya S., Shelton J.M., Bassel-Duby R., Olson E.N. Postnatal genome editing partially restores dystrophin expression in a mouse model of muscular dystrophy. Science. 2015; 351(6271):400-403.
72. Long C., McAnally J.R., Shelton J.M., Mireault A.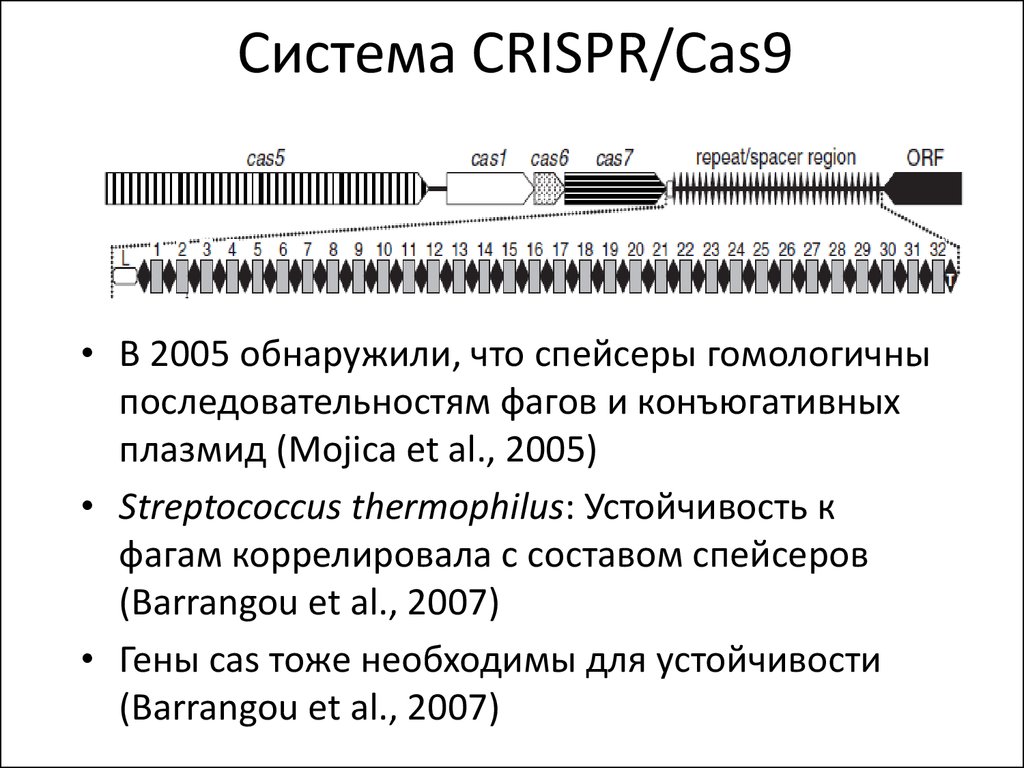 A., Bassel-Duby R., Olson E.N. Prevention of muscular dystrophy in mice by CRISPR/Cas9-mediated editing of germline DNA. Science. 2014; 345(6201):1184-1188.
A., Bassel-Duby R., Olson E.N. Prevention of muscular dystrophy in mice by CRISPR/Cas9-mediated editing of germline DNA. Science. 2014; 345(6201):1184-1188.
73. Makarova K.S., Wolf Y.I., Alkhnbashi O.S., Costa F., Shah S.A., Saunders S.J., Barrangou R., Brouns S.J.J., Charpentier E., Haft D.H., Horvath P., Moineau S., Mojica F.J.M., Terns R.M., Terns M.P., White M.F., Yakunin A.F., Garrett R.A., van der Oost J., Backofen R., Koonin E. V An updated evolutionary classification of CRISPRCas systems. Nat. Rev. Microbiol. 2015;13(11):722-736.
74. Mali P., Aach J., Stranges P.B., Esvelt K.M., Moosburner M., Kosuri S., Yang L., Church G.M. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat. Biotechnol. 2013;31(9):833-838.
75. Malina A., Cameron C.J.F., Robert F., Blanchette M., Dostie J., Pelletier J. PAM multiplicity marks genomic target sites as inhibitory to CRISPR-Cas9 editing. Nat. Commun. 2015;6:10124.
76. Mandal P.K., Ferreira L.M.R., Collins R., Meissner T.B., Boutwell C.L., Friesen M., Vrbanac V., Garrison B.S., Stortchevoi A., Bryder D., Musunuru K., Brand H., Tager A.M., Allen T.M., Talkowski M.E., Rossi D.J., Cowan C.A. Efficient ablation of genes in human hematopoietic stem and effector cells using CRISPR/Cas9. Cell Stem Cell. 2014;15(5):643-652.
77. Maruyama T., Dougan S.K., Truttmann M.C., Bilate A.M., Ingram J.R., Ploegh H.L. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining. Nat. Biotechnol. 2015;33(5):538-542.
78. Mendel G. Experiments in plant hybridization. J. Roy. Hortic. Soc. 1865;IV(1865):3-47.
79. Mora C., Tittensor D.P., Adl S., Simpson A.G.B., Worm B. How many species are there on Earth and in the ocean? PLoS Biol. 2011; 9(8):e1001127.
80. Nagano T., Fraser P. No-nonsense functions for long noncoding RNAs. Cell. 2011;145(2):178-181.
81. Naldini L. Gene therapy returns to centre stage. Nature. 2015;526 (7573):351-360.
Nature. 2015;526 (7573):351-360.
82. Nelson C.E., Gersbach C.A. Cas9 loosens its grip on off-target sites. Nat. Biotechnol. 2016;34(3):298-299.
83. Nelson C.E., Hakim C.H., Ousterout D.G., Thakore P.I., Moreb E.A., Rivera R.M.C., Madhavan S., Pan X., Ran F.A., Yan W.X., Asokan A., Zhang F., Duan D., Gersbach C.A. In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy. Science. 2015;351(6271):403-407.
84. Ni W., Qiao J., Hu S., Zhao X., Regouski M., Yang M., Polejaeva I.A., Chen C. Efficient gene knockout in goats using CRISPR/Cas9 system. PloS ONE. 2014;9(9):e106718.
85. Nishimasu H., Cong L., Yan W.X., Ran F.A., Zetsche B., Li Y., Kurabayashi A., Ishitani R., Zhang F., Nureki O. Crystal structure of Staphylococcus aureus Cas9. Cell. 2015;162(5):1113-1126.
86. Nishimasu H., Ran F.A., Hsu P.D., Konermann S., Shehata S.I., Dohmae N., Ishitani R., Zhang F., Nureki O. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 2014;156(5):935-949.
Cell. 2014;156(5):935-949.
87. Niu Y., Shen B., Cui Y., Chen Y., Wang J., Wang L., Kang Y., Zhao X., Si W., Li W., Xiang A.P., Zhou J., Guo X., Bi Y., Si C., Hu B., Dong G., Wang H., Zhou Z., Li T., Tan T., Pu X., Wang F., Ji S., Zhou Q., Huang X., Ji W., Sha J. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell. 2014;156(4): 836-843.
88. O’Connell M.R., Oakes B.L., Sternberg S.H., East-Seletsky A., Kaplan M., Doudna J.A. Programmable RNA recognition and cleavage by CRISPR/Cas9. Nature. 2014;516(7530):263-266.
89. Ota S., Hisano Y., Ikawa Y., Kawahara A. Multiple genome modifications by the CRISPR/Cas9 system in zebrafish. Genes to Cells. 2014;19(7):555-564.
90. Ousterout D.G., Kabadi A.M., Thakore P.I., Majoros W.H., Reddy T.E., Gersbach C.A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause Duchenne muscular dystrophy. Nat. Commun. 2015;6:6244.
91. Parnas O. , Jovanovic M., Eisenhaure T.M., Herbst R.H., Dixit A., Ye C.J., Przybylski D., Platt R.J., Tirosh I., Sanjana N.E., Shalem O., Satija R., Raychowdhury R., Mertins P., Carr S.A., Zhang F., Hacohen N., Regev A. A genome-wide CRISPR screen in primary immune cells to dissect regulatory networks. Cell. 2015;162(3):675-686.
, Jovanovic M., Eisenhaure T.M., Herbst R.H., Dixit A., Ye C.J., Przybylski D., Platt R.J., Tirosh I., Sanjana N.E., Shalem O., Satija R., Raychowdhury R., Mertins P., Carr S.A., Zhang F., Hacohen N., Regev A. A genome-wide CRISPR screen in primary immune cells to dissect regulatory networks. Cell. 2015;162(3):675-686.
92. Pattanayak V., Lin S., Guilinger J.P., Ma E., Doudna J.A., Liu D.R. High-throughput profiling of off-target DNA cleavage reveals RNAprogrammed Cas9 nuclease specificity. Nat. Biotechnol. 2013; 31(9):839-843.
93. Peng J., Wang Y., Jiang J., Zhou X., Song L., Wang L., Ding C., Qin J., Liu L., Wang W., Liu J., Huang X., Wei H., Zhang P. Production of human albumin in pigs through CRISPR/Cas9-mediated knockin of human cDNA into swine albumin locus in the zygotes. Sci. Reports. 2015;5:16705.
94. Pinder J., Salsman J., Dellaire G. Nuclear domain “knock-in” screen for the evaluation and identification of small molecule enhancers of CRISPR-based genome editing. Nucl. Acids Res. 2015;43(19): 9379-9392.
Nucl. Acids Res. 2015;43(19): 9379-9392.
95. Platt R.J., Chen S., Zhou Y., Yim M.J., Swiech L., Kempton H.R., Dahlman J.E., Parnas O., Eisenhaure T.M., Jovanovic M., Graham D.B., Jhunjhunwala S., Heidenreich M., Xavier R.J., Langer R., Anderson D.G., Hacohen N., Regev A., Feng G., Sharp P.A., Zhang F. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 2014;159(2):440-455.
96. Ran F.A., Cong L., Yan W.X., Scott D.A., Gootenberg J.S., Kriz A.J., Zetsche B., Shalem O., Wu X., Makarova K.S., Koonin E.V., Sharp P.A., Zhang F. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 2015;520(7546):186-191.
97. Ran F.A., Hsu P.D., Lin C.-Y., Gootenberg J.S., Konermann S., Trevino A.E., Scott D.A., Inoue A., Matoba S., Zhang Y., Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell. 2013;154(6):1380-1389.
98. Robert F., Barbeau M., Éthier S., Dostie J., Pelletier J. Pharmacological inhibition of DNA-PK stimulates Cas9-mediated genome editing. Gen. Med. 2015;7(1):93.
Gen. Med. 2015;7(1):93.
99. Schmid-Burgk J.L., Chauhan D., Schmidt T., Ebert T.S., Reinhardt J., Endl E., Hornung V. A Genome-wide CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) screen identifies NEK7 as an essential component of NLRP3 inflammasome activation. J. Biol. Chem. 2016;291(1):103-109.
100. Semenova E., Jore M.M., Datsenko K.A., Semenova A., Westra E.R., Wanner B., van der Oost J., Brouns S.J., Severinov K. Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence. Proc. Natl Acad. Sci. USA. 2011;108(25):10098-10103.
101. Shalem O., Sanjana N.E., Zhang F. High-throughput functional genomics using CRISPR-Cas9. Nat. Rev. Genet. 2015;16(5):299-311.
102. Shechner D.M., Hacisuleyman E., Younger S.T., Rinn J.L. Multiplexable, locus-specific targeting of long RNAs with CRISPR-Display. Nat. Methods. 2015;12(7):664-670.
103. Slaymaker I.M., Gao L., Zetsche B., Scott D.A., Yan W. X., Zhang F. Rationally engineered Cas9 nucleases with improved specificity. Science. 2015;351(6268):84-88.
X., Zhang F. Rationally engineered Cas9 nucleases with improved specificity. Science. 2015;351(6268):84-88.
104. Song Y., Yuan L., Wang Y., Chen M., Deng J., Lv Q., Sui T., Li Z., Lai L. Efficient dual sgRNA-directed large gene deletion in rabbit with CRISPR/Cas9 system. Cell. Mol. Life Sci. 2016;1:1-10.
105. Sternberg S.H., Redding S., Jinek M., Greene E.C., Doudna J.A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 2014;507(7490):62-67.
106. Tabebordbar M., Zhu K., Cheng J.K.W., Chew W.L., Widrick J.J., Yan W.X., Maesner C., Wu E.Y., Xiao R., Ran F.A., Cong L., Zhang F., Vandenberghe L.H., Church G.M., Wagers A.J., Vandenberghe H., Church G.M., Wagers A.J. In vivo gene editing in dystrophic mouse muscle and muscle stem cells. Science. 2015;351(6271):407-411.
107. Tan W., Proudfoot C., Lillico S.G., Whitelaw C.B.A. Gene targeting, genome editing: from Dolly to editors. Transgenic Res. 2016;25(3): 273-287.
108. Tanenbaum M.E., Gilbert L. A., Qi L.S., Weissman J.S., Vale R.D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 2014;159(3):635-646.
A., Qi L.S., Weissman J.S., Vale R.D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 2014;159(3):635-646.
109. Tebas P., Stein D., Tang W.W., Frank I., Wang S.Q., Lee G., Spratt S.K., Surosky R.T., Giedlin M.A., Nichol G., Holmes M.C., Gregory P.D., Ando D.G., Kalos M., Collman R.G., Binder-Scholl G., Plesa G., Hwang W.-T., Levine B.L., June C.H. Gene editing of CCR5 in autologous CD4 T cells of persons infected with HIV. New Engl. J. Med. 2014;370(10):901-910.
110. Toledo C.M., Ding Y., Hoellerbauer P., Davis R.J., Basom R., Girard E.J., Lee E., Corrin P., Hart T., Bolouri H., Davison J., Zhang Q., Hardcastle J., Aronow B.J., Plaisier C.L., Baliga N.S., Moffat J., Lin Q., Li X.-N., Nam D.-H., Lee J., Pollard S.M., Zhu J., Delrow J.J., Clurman B.E., Olson J.M., Paddison P.J. Genome-wide CRISPR-Cas9 screens reveal loss of redundancy between PKMYT1 and WEE1 in glioblastoma Stem-like Cells. Cell Reports. 2015;13(11):2425-2439.
111. Tsai S.Q., Wyvekens N., Khayter C., Foden J.A., Thapar V., Reyon D., Goodwin M.J., Aryee M.J., Joung J.K. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. Nat. Biotechnol. 2014;32(6):569-576.
112. Unckless R.L., Messer P.W., Connallon T., Clark A.G. Modeling the manipulation of natural populations by the mutagenic chain reaction. Genetics. 2015;201(2):425-431.
113. Wang H., Yang H., Shivalila C.S., Dawlaty M.M., Cheng A.W., Zhang F., Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/cas-mediated genome engineering. Cell. 2013;153(4):910-918.
114. Wang X., Zhou J., Cao C., Huang J., Hai T., Wang Y., Zheng Q., Zhang H., Qin G., Miao X., Wang H., Cao S., Zhou Q., Zhao J. Efficient CRISPR/Cas9-mediated biallelic gene disruption and sitespecific knockin after rapid selection of highly active sgRNAs in pigs. Sci. Reports. 2015a;5:13348.
115. Wang Y., Zhang Z.T., Seo S.O., Choi K., Lu T., Jin Y. S., Blaschek H.P. Markerless chromosomal gene deletion in Clostridium beijerinckii using CRISPR/Cas9 system. J. Biotechnol. 2015b;200:1-5.
S., Blaschek H.P. Markerless chromosomal gene deletion in Clostridium beijerinckii using CRISPR/Cas9 system. J. Biotechnol. 2015b;200:1-5.
116. Whitworth K.M., Lee K., Benne J.A., Beaton B.P., Spate L.D., Murphy S.L., Samuel M.S., Mao J., O’Gorman C., Walters E.M., Murphy C.N., Driver J., Mileham A., McLaren D., Wells K.D., Prather R.S. Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos. Biol. Reprod. 2014;91(3):78.
117. Wiedenheft B., Lander G.C., Zhou K., Jore M.M., Brouns S.J., van der Oost J., Doudna J.A., Nogales E. Structures of the RNA-guided surveillance complex from a bacterial immune system. Nature. 2011; 477(7365):486-489.
118. Wijshake T., Baker D.J., van de Sluis B. Endonucleases: new tools to edit the mouse genome. Biochim. Bioph. Acta. 2014;1842(10): 1942-1950.
119. Wright A.V., Nunez J.K., Doudna J.A. Review biology and applications of CRISPR systems: harnessing nature’s toolbox for genome engineering. Cell. 2016;164(1-2):29-44.
Cell. 2016;164(1-2):29-44.
120. Wright A.V., Sternberg S.H., Taylor D.W., Staahl B.T., Bardales J.A., Kornfeld J.E., Doudna J.A. Rational design of a split-Cas9 enzyme complex. Proc. Natl Acad. Sci. USA. 2015;112(10):2984-2989.
121. Wu B., Luo L., Gao X.J. Cas9-triggered chain ablation of cas9 as a gene drive brake. Nat. Biotechnol. 2016;34(2):137-138.
122. Wu X., Scott D.A., Kriz A.J., Chiu A.C., Hsu P.D., Dadon D.B., Cheng A.W., Trevino A.E., Konermann S., Chen S., Jaenisch R., Zhang F., Sharp P.A. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 2014;32(7):670-676.
123. Wu Y., Liang D., Wang Y., Bai M., Tang W., Bao S., Yan Z., Li D., Li J. Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell. 2013;13(6):659-662.
124. Wyvekens N., Topkar V.V., Khayter C., Joung J.K., Tsai S.Q. Dimeric CRISPR RNA-guided FokI-dCas9 nucleases directed by truncated gRNAs for highly specific genome editing. Hum. Gene Ther. 2015; 26(7):425-431.
Gene Ther. 2015; 26(7):425-431.
125. Xiao A., Wang Z., Hu Y., Wu Y., Luo Z., Yang Z., Zu Y., Li W., Huang P., Tong X., Zhu Z., Lin S., Zhang B. Chromosomal deletions and inversions mediated by TALENs and CRISPR/Cas in zebrafish. Nucl. Acids Res. 2013;41(14):1-11.
126. Xue H.Y., Ji L.J., Gao A.M., Liu P., He J.D., Lu X.J. CRISPR-Cas9 for medical genetic screens: applications and future perspectives. J. Med. Genet. 2016;53(2):91-97.
127. Yang D., Scavuzzo M.A., Chmielowiec J., Sharp R., Bajic A., Borowiak M. Enrichment of G2/M cell cycle phase in human pluripotent stem cells enhances HDR-mediated gene repair with customizable endonucleases. Sci. Reports. 2016;6:21264.
128. Yang H., Wang H., Shivalila C.S., Cheng A.W., Shi L., Jaenisch R. Onestep generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 2013a;154(6): 1370-1379.
129. Yang L., Grishin D., Wang G., Aach J., Zhang C.-Z., Chari R., Homsy J., Cai X. , Zhao Y., Fan J.-B., Seidman C., Seidman J., Pu W., Church G. Targeted and genome-wide sequencing reveal single nucleotide variations impacting specificity of Cas9 in human stem cells. Nat. Commun. 2014;5:5507.
, Zhao Y., Fan J.-B., Seidman C., Seidman J., Pu W., Church G. Targeted and genome-wide sequencing reveal single nucleotide variations impacting specificity of Cas9 in human stem cells. Nat. Commun. 2014;5:5507.
130. Yang L., Guell M., Byrne S., Yang J.L., De Los Angeles A., Mali P., Aach J., Kim-Kiselak C., Briggs A.W., Rios X., Huang P.Y., Daley G., Church G. Optimization of scarless human stem cell genome editing. Nucl. Acids Res. 2013b;41(19):9049-9061.
131. Yang L., Guell M., Niu D., George H., Lesha E., Grishin D., Aach J., Shrock E., Xu W., Poci J., Cortazio R., Wilkinson R.A., Fishman J.A., Church G. Genome-wide inactivation of porcine endogenous retroviruses (PERVs). Science. 2015;350(6264):1101-1104.
132. Ye L., Wang J., Beyer A.I., Teque F., Cradick T.J., Qi Z., Chang J.C., Bao G., Muench M.O., Yu J., Levy J.A., Kan Y.W. Seamless modification of wild-type induced pluripotent stem cells to the natural CCR5Δ32 mutation confers resistance to HIV infection. Proc. Natl Acad. Sci. USA. 2014;111(26):9591-9596.
Natl Acad. Sci. USA. 2014;111(26):9591-9596.
133. Yen S.T., Zhang M., Deng J.M., Usman S.J., Smith C.N., Parker-Thornburg J., Swinton P.G., Martin J.F., Behringer R.R. Somatic mosaicism and allele complexity induced by CRISPR/Cas9 RNA injections in mouse zygotes. Dev. Biol. 2014;393(1):3-9.
134. Yin H., Xue W., Chen S., Bogorad R.L., Benedetti E., Grompe M., Koteliansky V., Sharp P.A., Jacks T., Anderson D.G. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype. Nat. Biotechnol. 2014;32(6):551-553.
135. Young C.S., Hicks M.R., Ermolova N.V., Nakano H., Jan M., Younesi S., Karumbayaram S., Kumagai-Cresse C., Wang D., Zack J.A., Kohn D.B., Nakano A., Nelson S.F., Miceli M.C., Spencer M.J., Pyle A.D. A Single CRISPR-Cas9 deletion strategy that targets the majority of DMD patients restores dystrophin function in hiPSCderived muscle cells. Cell Stem Cell. 2016;18(4)533-540.
136. Yu C., Liu Y., Ma T., Liu K., Xu S., Zhang Y., Liu H., La Russa M.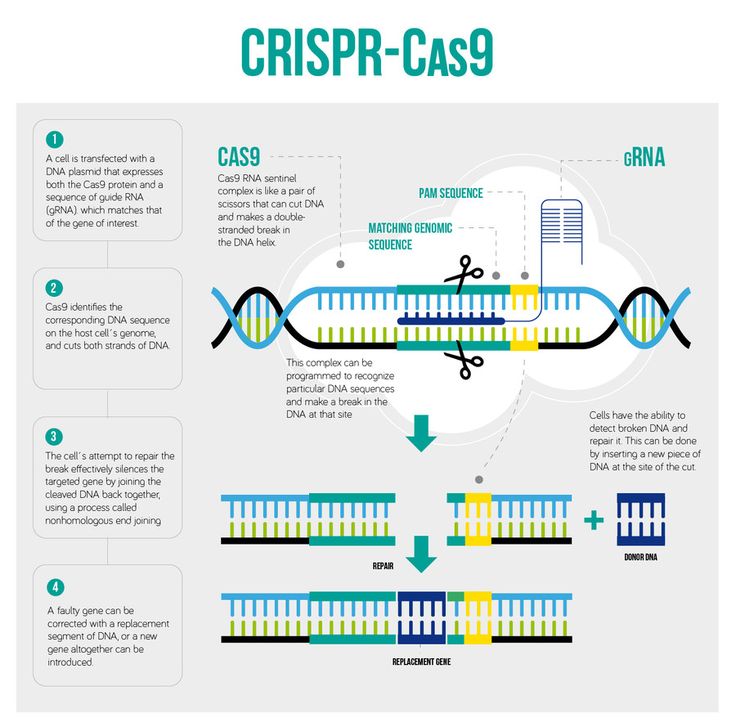 , Xie M., Ding S., Qi L.S. Small molecules enhance CRISPR genome editing in pluripotent stem cells. Cell Stem Cell. 2015;16(2): 142-147.
, Xie M., Ding S., Qi L.S. Small molecules enhance CRISPR genome editing in pluripotent stem cells. Cell Stem Cell. 2015;16(2): 142-147.
137. Yuan L., Sui T., Chen M., Deng J., Huang Y., Zeng J., Lv Q., Song Y., Li Z., Lai L. CRISPR/Cas9-mediated GJA8 knockout in rabbits recapitulates human congenital cataracts. Sci. Reports. 2016;6: 22024.
138. Zalatan J.G., Lee M.E., Almeida R., Gilbert L.A., Whitehead E.H., La Russa M., Tsai J.C., Weissman J.S., Dueber J.E., Qi L.S., Lim W.A. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 2014;160(1-2):339-350.
139. Zetsche B., Gootenberg J.S., Abudayyeh O.O., Slaymaker I.M., Makarova K.S., Essletzbichler P., Volz S.E., Joung J., van der Oost J., Regev A., Koonin E.V., Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 2015a;163(3): 759-771.
140. Zetsche B., Volz S.E., Zhang F. A split-Cas9 architecture for inducible genome editing and transcription modulation. Nat. Biotechnol. 2015b;33(2):139-142.
Nat. Biotechnol. 2015b;33(2):139-142.
141. Zhang L., Jia R., Palange N.J., Satheka A.C., Togo J., An Y., Humphrey M., Ban L., Ji Y., Jin H., Feng X., Zheng Y. Large genomic fragment deletions and insertions in mouse using CRISPR/Cas9. PLoS ONE. 2015;10(3):1-14.
142. Zheng Q., Cai X., Tan M.H., Schaffert S., Arnold C.P., Gong X., Chen C.Z., Huang S. Precise gene deletion and replacement using the CRISPR/Cas9 system in human cells. BioTechniques. 2014; 57(3):115-124.
143. Zhou J., Wang J., Shen B., Chen L., Su Y., Yang J., Zhang W., Tian X., Huang X. Dual sgRNAs facilitate CRISPR/Cas9-mediated mouse genome targeting. FEBS J. 2014;281(7):1717-1725.
Что такое CRISPR-Cas9? – Ваш геном
Что такое CRISPR-Cas9?
- CRISPR-Cas9 — это уникальная технология, которая позволяет генетикам и исследователям-медикам редактировать части генома, удаляя, добавляя или изменяя участки последовательности ДНК.
- В настоящее время это самый простой, универсальный и точный метод генетических манипуляций, поэтому он вызывает ажиотаж в научном мире.

Как это работает?
- CRISPR-Cas9система состоит из двух ключевых молекул, которые вносят изменение (мутацию) в ДНК. Это:
- фермент под названием Cas9. Это действует как пара «молекулярных ножниц», которые могут разрезать две нити ДНК в определенном месте генома, чтобы затем можно было добавлять или удалять фрагменты ДНК.
- фрагмент РНК, называемый направляющей РНК (гРНК). Он состоит из небольшого фрагмента предварительно сконструированной последовательности РНК (длиной около 20 оснований), расположенного внутри более длинного каркаса РНК. Каркасная часть связывается с ДНК, а предварительно разработанная последовательность «направляет» Cas9.в правую часть генома. Это гарантирует, что фермент Cas9 врезается в нужную точку генома.
- Направляющая РНК предназначена для поиска и связывания определенной последовательности ДНК. Направляющая РНК имеет основания РНК, комплементарные основаниям целевой последовательности ДНК в геноме.
 Это означает, что, по крайней мере теоретически, направляющая РНК будет связываться только с последовательностью-мишенью и никакими другими областями генома.
Это означает, что, по крайней мере теоретически, направляющая РНК будет связываться только с последовательностью-мишенью и никакими другими областями генома. - Cas9 следует за направляющей РНК в то же место в последовательности ДНК и делает разрез по обеим цепям ДНК.
- На этом этапе клетка распознает, что ДНК повреждена, и пытается ее восстановить.
- Ученые могут использовать механизм репарации ДНК для внесения изменений в один или несколько генов в геном интересующей клетки.
Схема, показывающая, как работает инструмент редактирования CRISPR-Cas9.
Изображение предоставлено: Genome Research Limited.
Как он был разработан?
- Некоторые бактерии имеют встроенную систему редактирования генов, аналогичную CRISPR-Cas9система, которую они используют для реагирования на вторгающиеся патогены, такие как вирусы, очень похожа на иммунную систему.
- Используя CRISPR, бактерии вырезают части вирусной ДНК и оставляют часть ее позади, чтобы помочь им распознать вирус и защититься от него в следующий раз, когда он атакует.

- Ученые адаптировали эту систему, чтобы ее можно было использовать в других клетках животных, включая мышей и людей.
Какие еще существуют методы изменения генов?
- На протяжении многих лет ученые изучали генетику и функции генов, изучая последствия изменений в ДНК.
- Если вы можете создать изменение в гене, либо в клеточной линии, либо во всем организме, можно затем изучить эффект этого изменения, чтобы понять, какова функция этого гена.
- Долгое время генетики использовали химические вещества или радиацию, чтобы вызывать мутации. Однако у них не было возможности контролировать, где в геноме произойдет мутация.
- В течение нескольких лет ученые использовали «нацеливание на гены» для внесения изменений в определенные участки генома путем удаления или добавления либо целых генов, либо отдельных оснований.
- Традиционное нацеливание на гены оказалось очень ценным для изучения генов и генетики, однако для создания мутации требуется много времени и это довольно дорого.

- Недавно было разработано несколько технологий «редактирования генов» для улучшения методов нацеливания на гены, включая системы CRISPR-Cas, эффекторные нуклеазы, подобные активаторам транскрипции (TALEN) и нуклеазы с цинковыми пальцами (ZFN).
- Система CRISPR-Cas9 в настоящее время выделяется как самая быстрая, дешевая и самая надежная система для «редактирования» генов.
Каковы приложения и последствия?
- CRISPR-Cas9 обладает большим потенциалом в качестве инструмента для лечения ряда заболеваний с генетическим компонентом, включая рак, гепатит В или даже высокий уровень холестерина.
- Многие из предлагаемых приложений включают редактирование геномов соматических (не репродуктивных) клеток, но существует большой интерес и споры о возможности редактирования зародышевых (репродуктивных) клеток.
- Поскольку любые изменения, происходящие в клетках зародышевой линии, будут передаваться из поколения в поколение, это имеет важные этические последствия.

- Проведение редактирования генов в клетках зародышевой линии в настоящее время является незаконным в Великобритании и большинстве других стран.
- Напротив, использование CRISPR-Cas9 и других технологий редактирования генов в соматических клетках не вызывает сомнений. Действительно, они уже использовались для лечения заболеваний человека в небольшом числе исключительных и/или опасных для жизни случаев.
Сперматозоид и яйцеклетка. Проведение редактирования генов в клетках зародышевой линии в настоящее время является незаконным в Великобритании.
Изображение предоставлено Shutterstock
Каково будущее CRISPR-Cas9?
- Скорее всего, пройдет много лет, прежде чем CRISPR-Cas9 станет рутинно использоваться у людей.
- Многие исследования по-прежнему сосредоточены на его использовании на животных моделях или изолированных клетках человека с целью в конечном итоге использовать эту технологию для рутинного лечения заболеваний у людей.

- Проводится много работы по устранению «нецелевых» эффектов, когда система CRISPR-Cas9 врезается в ген, отличный от того, который предназначался для редактирования.
Лучшее нацеливание на CRISPR-Cas9
- В большинстве случаев направляющая РНК состоит из определенной последовательности из 20 оснований. Они комплементарны целевой последовательности в редактируемом гене. Однако не все 20 оснований должны совпадать, чтобы направляющая РНК могла связываться.
- Проблема в том, что последовательность, например, с 19 из 20 комплементарных оснований, может существовать где-то в совершенно другом месте генома. Это означает, что направляющая РНК может связываться с ней вместо последовательности-мишени или так же, как и с последовательностью-мишенью.
- Затем фермент Cas9 будет разрезаться не в том месте и, в конечном итоге, вызовет мутацию в неправильном месте. Хотя эта мутация может вообще не иметь значения для человека, она может повлиять на ключевой ген или другую важную часть генома.

- Ученые стремятся найти способ гарантировать точное связывание и разрезание CRISPR-Cas9. Этого можно достичь двумя способами:
- разработка лучших, более специфичных направляющих РНК с использованием наших знаний о последовательности ДНК генома и «нецелевом» поведении различных версий Cas9-комплекс гРНК.
- использование фермента Cas9, который разрезает только одну цепь ДНК-мишени, а не двойную. Это означает, что два фермента Cas9 и две направляющие РНК должны находиться в одном и том же месте, чтобы разрез был сделан. Это снижает вероятность того, что разрез будет сделан не в том месте.
Эта страница последний раз обновлялась 08 февраля 2022 г.
CRISPR/Cas9 и целевое редактирование генома: новая эра в молекулярной биологии
Разработка эффективных и надежных способов внесения точных и целенаправленных изменений в геном живых клеток является давней целью
для биомедицинских исследователей. Недавно появился новый инструмент на основе бактериального CRISPR-ассоциированного белка-9. нуклеаза (Cas9) из Streptococcus
нуклеаза (Cas9) из Streptococcus
pyogenes вызвал значительный ажиотаж (1). Это следует за несколькими попытками на протяжении многих лет манипулировать функцией генов, в том числе
гомологичная рекомбинация (2) и РНК-интерференция (РНКи) (3). РНК-интерференция, в частности, стала основным лабораторным продуктом, позволяющим делать недорогие
и высокопроизводительный опрос функции генов (4, 5), но он затруднен, поскольку обеспечивает только временное ингибирование функции генов
и непредсказуемые нецелевые эффекты (6). Другие недавние подходы к целенаправленной модификации генома — нуклеазы с цинковыми пальцами [ZFN, (7)] и
эффекторные нуклеазы, подобные активаторам транскрипции [TALENs (8)] – позволяют исследователям генерировать постоянные мутации путем введения двухцепочечных
перерывы, чтобы активировать пути восстановления. Эти подходы являются дорогостоящими и трудоемкими для разработки, что ограничивает их широкое использование.
особенно для крупномасштабных, высокопроизводительных исследований.
Что такое CRISPR/Cas9?
Функции CRISPR (регулярная кластеризация
Короткие палиндромные повторы с промежутками) и
CRISPR-ассоциированные (Cas) гены необходимы для
адаптивный иммунитет у некоторых бактерий и архей,
позволяет организмам реагировать на
устранить вторгшийся генетический материал. Эти повторы
были первоначально обнаружены в 1980-х годах в E. coli (9), но их функция не была подтверждена
до 2007 года Баррангоу и его коллеги, которые
показал, что S. thermophilus может приобретать устойчивость
против бактериофага путем интеграции
фрагмент генома инфекционного вируса в его
Локус CRISPR (10).
Три типа механизмов CRISPR были
идентифицированы, из которых тип II является наиболее изученным. В
в этом случае вторжение в ДНК вирусов или плазмид
разрезается на мелкие фрагменты и включается в
локус CRISPR среди серии коротких повторов
(около 20 б.п.). Локусы транскрибируются, и
транскрипты затем обрабатываются для создания небольших
РНК (crRNA – CRISPR RNA), которые используются для управления эффекторными эндонуклеазами, нацеленными на
вторжение в ДНК на основе комплементарности последовательностей
(Рисунок 1) (11).
Рисунок 1. Cas9
in vivo : бактериальный адаптивный иммунитет
На этапе приобретения чужеродная ДНК встраивается в бактериальный геном в локусах CRISPR. Затем локусы CRISPR транскрибируются и процессируются в crРНК во время биогенеза crРНК. Во время интерференции эндонуклеаза Cas9 в комплексе с crРНК и отдельной tracrРНК расщепляет чужеродную ДНК, содержащую 20-нуклеотидную комплементарную последовательность crРНК, примыкающую к последовательности PAM. (Рисунок выполнен не в масштабе.)
Один белок Cas, Cas9 (также известный как Csn1),
было показано, через нокдаун и спасение
эксперименты, чтобы стать ключевым игроком в определенных CRISPR
механизмы (в частности, системы CRISPR типа II).
Механизм CRISPR типа II уникален по сравнению с
другим системам CRISPR, так как только один белок Cas
(Cas9) необходим для молчания генов (12). В типе
II, Cas9 участвует в процессинге
crРНК (12) и отвечает за разрушение
ДНК-мишень (11). Функция Cas9 в обоих этих
шагов зависит от наличия двух нуклеазных доменов,
домен RuvC-подобной нуклеазы, расположенный в аминогруппе
конце и HNH-подобный домен нуклеазы, который находится
в средней области белка (13).
Для достижения сайт-специфического распознавания ДНК и
расщепления, Cas9 должен образовывать комплекс как с
crРНК и отдельная трансактивирующая crРНК
(tracrRNA или trRNA), частично комплементарная
к crРНК (11). Требуется tracrRNA
для созревания crРНК из первичного транскрипта
кодирующие несколько пре-crРНК. Это происходит в
наличие РНКазы III и Cas9 (12).
При разрушении ДНК-мишени HNH
и RuvC-подобные нуклеазные домены разрезают обе ДНК
нити, образующие двухцепочечные разрывы (DSB)
в сайтах, определяемых целевой последовательностью из 20 нуклеотидов
в ассоциированном транскрипте crRNA (11, 14).
Домен HNH расщепляет комплементарный
цепь, в то время как домен RuvC расщепляет некомплементарную
прядь.
Активность двухцепочечной эндонуклеазы Cas9
также требует, чтобы короткая консервативная последовательность (2–5
nts), известный как мотив, ассоциированный с протоспейсером (PAM),
следует сразу за 3′- комплементарной crРНК
последовательность (15). На самом деле, даже полностью дополняющие
последовательности игнорируются Cas9-РНК в
отсутствие последовательности PAM (16).
Cas9 и CRISPR как новый инструмент в
Молекулярная биология
Простота нуклеазы CRISPR типа II,
только с тремя обязательными компонентами (Cas9вместе
с crRNA и trRNA) делает эту систему
поддается адаптации для редактирования генома. Этот
потенциал был реализован в 2012 году компанией Doudna
и лаборатории Шарпантье (11). По типу II
Система CRISPR, описанная ранее авторами
разработал упрощенную двухкомпонентную систему
путем объединения тРНК и кРНК в одну
синтетическая одиночная направляющая РНК (sgRNA). sgRNA запрограммированный
Cas9 оказался столь же эффективным
как Cas9, запрограммированный с отдельными тРНК и
crRNA в управлении целевыми изменениями генов
(Рисунок 2А).
На сегодняшний день три разных варианта Cas9
нуклеаза была принята в редактировании генома
протоколы. Первым является Cas9 дикого типа, который
может сайт-специфически расщеплять двухцепочечные
ДНК, что приводит к активации двухцепочечной
поломка (DSB) ремонт техники. DSB могут
репарироваться клеточным негомологичным
Путь End Joining (NHEJ) (17), в результате
во вставках и/или делециях (вставках), которые
нарушить целевой локус. В качестве альтернативы, если донорная матрица гомологична целевому
В качестве альтернативы, если донорная матрица гомологична целевому
locus поставляется, DSB может быть отремонтирован
путь гомологически направленной репарации (HDR)
позволяет точно замещать мутации
(рис. 2А) (17, 18).
Конг и его коллеги (1) взяли систему Cas9
еще один шаг к повышению точности за счет
разработка мутантной формы, известной как Cas9D10A,
только с никазной активностью. Это означает, что он расщепляется
только одна цепь ДНК и не активирует
NHEJ. Вместо этого при наличии гомологичного
шаблон репарации, репарация ДНК проводится через
только путь HDR с высокой точностью, что приводит к
редуцированные мутации indel (1, 11, 19). Cas9D10A это
еще более привлекательным с точки зрения целевой специфичности
когда локусы нацелены на парный Cas9комплексы
предназначен для создания соседних разрывов ДНК (20) (см.
дополнительные подробности о «парных никазах» на рисунке 2B).
Третий вариант – дефицитный по нуклеазе Cas9.
(dCas9, рис. 2C) (21). Мутации H840A в
Домен HNH и D10A в домене RuvC
инактивируют активность расщепления, но не предотвращают
Связывание ДНК (11, 22).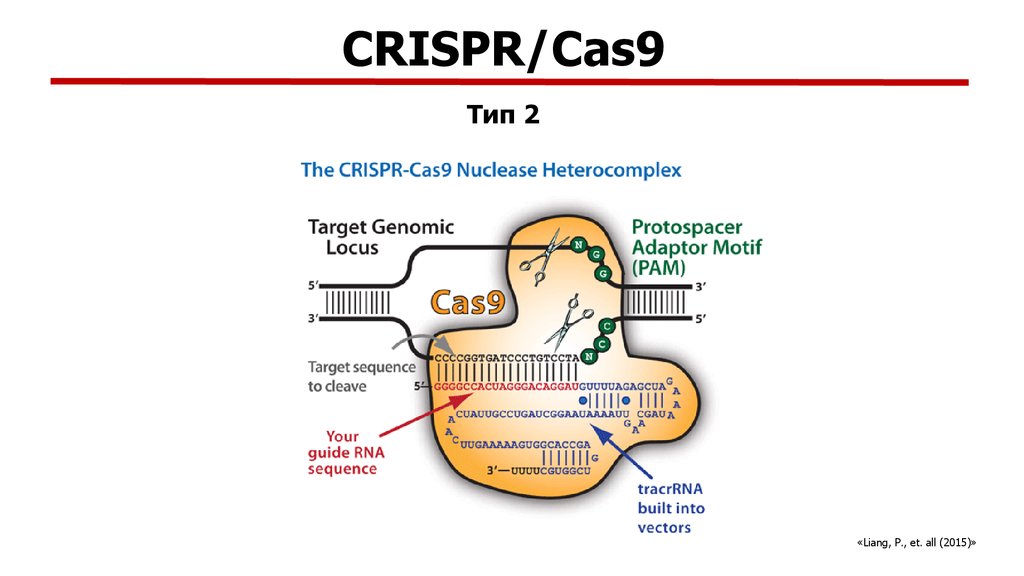 Поэтому этот вариант
Поэтому этот вариант
может использоваться для специфического нацеливания на любую последовательность
участок генома без расщепления. Вместо этого путем слияния с различными эффекторными доменами dCas9может использоваться как для подавления гена, так и для активации
инструмент (21, 23–26). Кроме того, его можно использовать
как средство визуализации. Например, Чен и
коллеги использовали dCas9, слитый с Enhanced Green
Флуоресцентный белок (EGFP) для визуализации повторяющихся
Последовательности ДНК с одиночной sgRNA или неповторяющимися
локусы с использованием нескольких sgRNAs (27).
Рис. 2. Системные приложения CRISPR/Cas9
А. Сайт нуклеазы Cas9 дикого типа специфически расщепляет двухцепочечную ДНК, активируя механизм репарации двухцепочечных разрывов. В отсутствие гомологичного шаблона репарации негомологичное соединение концов может привести к вставкам, нарушающим последовательность-мишень. В качестве альтернативы, точные мутации и нокауты могут быть сделаны путем предоставления гомологичной матрицы репарации и использования пути репарации, направленного на гомологию.
B. Мутированный Cas9 создает сайт-специфический одноцепочечный разрыв. Две sgRNA можно использовать для введения двухцепочечного разрыва, расположенного в шахматном порядке, который затем может подвергаться направленной по гомологии репарации.
C. Дефицитный по нуклеазе Cas9 может быть слит с различными эффекторными доменами, что обеспечивает специфическую локализацию. Например, активаторы транскрипции, репрессоры и флуоресцентные белки.
Эффективность прицеливания и нецелевое попадание
Мутации
Эффективность таргетинга, или процент желаемого
достигнутая мутация, является одним из наиболее важных
параметры, по которым можно оценить редактирование генома
инструмент. Целевая эффективность Cas9сравнивает
выгодно с более устоявшимися методами, такими
как TALEN или ZFN (8). Например, у человека
ячейки, специально разработанные ZFN и TALEN могут
достичь КПД в диапазоне от 1% до
50% (29–31). Напротив, система Cas9 имеет
сообщалось, что эффективность достигает >70%
у рыбок данио (32) и растений (33), а также
от 2 до 5% в индуцированных плюрипотентных стволовых клетках (34).
Кроме того, Чжоу и его коллеги смогли
улучшить нацеливание на геном до 78% в одноклеточном
эмбрионов мышей и получили эффективную зародышевую линию
передача с помощью двойных sgRNAs
одновременно нацелены на отдельный ген (35).
Широко используемым методом выявления мутаций является
Анализ обнаружения мутаций эндонуклеазы I T7 (36,
37) (рис. 3). Этот анализ обнаруживает гетеродуплекс
ДНК, полученная в результате отжига ДНК
цепь, включая желаемые мутации, с диким типом
Цепь ДНК (37).
Рисунок 3. Анализ эффективности нацеливания на эндонуклеазу I T7
Геномная ДНК амплифицируется с помощью праймеров, заключающих в скобки модифицированный локус. Затем продукты ПЦР денатурируют и повторно отжигают, получая 3 возможные структуры. Дуплексы, содержащие несоответствие, расщепляются эндонуклеазой I Т7. Затем ДНК разделяют электрофоретически, и для расчета эффективности нацеливания используют анализ фрагментов.
Другим важным параметром является частота
нецелевые мутации. Такие мутации, вероятно,
Такие мутации, вероятно,
появляются на сайтах, которые отличаются лишь несколькими
нуклеотидов по сравнению с исходной последовательностью,
пока они находятся рядом с последовательностью PAM.
Это происходит, поскольку Cas9 может переносить до 5 оснований.
несовпадения в области протоспейсера (36) или
разница в единственном основании в последовательности PAM (38).
Нецелевые мутации, как правило, сложнее
обнаруживать, требуя секвенирования всего генома
их полностью.
Последние улучшения системы CRISPR для
было сделано уменьшение нецелевых мутаций
с помощью укороченной гРНК (truncated
в последовательности, полученной из crРНК), или путем добавления
два дополнительных нуклеотида гуанина (G) к 5′-концу
(28, 37). Другой способ, которым пытались воспользоваться исследователи
свести к минимуму нецелевые эффекты с использованием
«парные никасы» (20). Эта стратегия использует D10A
Cas9 и две sgRNA, комплементарные
прилегающая область на противоположных нитях целевого сайта
(Рисунок 2Б). Хотя это вызывает DSB в
Хотя это вызывает DSB в
ДНК-мишени, ожидается, что она создаст только одиночные разрывы в нецелевых местах и, следовательно, приведет к
с минимальными нецелевыми мутациями.
Путем использования вычислений для уменьшения нецелевого
мутаций, несколько групп разработали
инструменты, облегчающие идентификацию
потенциальные целевые сайты CRISPR и оценить их
возможность нецелевого расщепления. Примеры
включают CRISPR Design Tool (38) и
ZiFiT Targeter, версия 4.2 (39, 40).
приложения в качестве редактирования генома
и Инструмент определения генома
После первой демонстрации в 2012 г.
(9), система CRISPR/Cas9 получила широкое распространение.
усыновленный. Это уже успешно используется
для нацеливания на важные гены во многих клеточных линиях и
организмы, включая человека (34), бактерии (41),
данио (32), C. elegans (42), растения (34), Xenopus
tropicalis (43), дрожжи (44), Drosophila (45), обезьяны
(46), кролики (47), свиньи (42), крысы (48) и мыши
(49). Несколько групп уже воспользовались
Несколько групп уже воспользовались
этот метод для введения одноточечных мутаций
(удаления или вставки) в конкретную цель
ген через одну гРНК (14, 21, 29). Используя
пара гРНК-направленных нуклеаз Cas9 вместо этого,
также возможно вызвать большие делеции или
геномные перестройки, такие как инверсии
или транслокации (50). Недавнее захватывающее
разработка заключается в использовании dCas9версия
системы CRISPR/Cas9 для нацеливания на белок
домены для регуляции транскрипции (26, 51,
52), эпигенетическая модификация (25) и микроскопический
визуализация специфических локусов генома (27).
Система CRISPR/Cas9 требует только доработки
crРНК для изменения специфичности мишени.
Это контрастирует с другими инструментами редактирования генома,
в том числе цинковый палец и ТАЛЕНы, где редизайн
требуется интерфейс белок-ДНК.
Кроме того, CRISPR/Cas9 позволяет быстро
полногеномный опрос функции генов
путем создания больших библиотек гРНК (51, 53) для
геномный скрининг.
Будущее CRISPR/Cas9
Быстрый прогресс в превращении Cas9 в набор
инструментов для исследований в области клеточной и молекулярной биологии
был замечательным, вероятно, из-за простоты,
высокая эффективность и универсальность системы. Принадлежащий
Принадлежащий
дизайнерские нуклеазные системы, доступные в настоящее время для
прецизионная инженерия генома, CRISPR/Cas
система на сегодняшний день является наиболее удобной для пользователя. Сейчас
также ясно, что потенциал Cas9 выходит за рамки
Расщепление ДНК и его полезность для генома
Локус-специфическое рекрутирование белков, вероятно,
ограничиваться только нашим воображением.
Посмотреть продукты NEB для редактирования генома
Каталожные номера
- Конг Л. и др. (2013) Наука , 339, 819–823.
- Capecchi, MR (2005) Nat. Преподобный Жене. 6, 507–512.
- Файр, А., и др. (1998) Природа, 391, 806–811.
- Эльбашир С.Мм и др. (2002) Методы , 26, 199–213.
- Мартинес, Дж., и др. (2003) Nucleic Acids Res. Доп. 333.
- Алик, Н., и др. (2012) PLoS One, 7, e45367.
- Миллер, Дж. и др. (2005) Мол. тер. 11, С35–С35.

- Муссолино, К., и др. (2011) Nucleic Acids Res. 39, 9283–9293.
- Ишино Ю. и др. (1987) J. Bacteriol. 169, 5429–5433.
- Баррангу Р. и др. (2007). Наука, 315, 1709–1712.
- Джинек М. и др. (2012) Наука, 337, 816–821.
- Делчева Е. и др. (2011) Природа, 471, 602–607.
- Сапранаускас Р. и др. (2011) Nucleic Acids Res. 39, 9275–9282.
- Нисимасу Х. и др. (2014) Cell, doi:10.1016/j.cell.2014.02.001
- Суортс, округ Колумбия, и др. (2012) PLoS One, 7:e35888.
- Штернберг С.Х. и др. (2014) Nature, doi:10.1038/nature13011.
- Overballe-Petersen, S., et al. (2013) Proc. Натл. акад. науч. США 110, 19860–19865.
- Гонг С. и др. (2005) Нац. Структура Мол. биол. 12, 304–312.
- Дэвис, Л., Майзелс, Н. (2014) Proc. Натл. акад. науч. США, 111, E924–932.
- Ран Ф.
 А. и др. (2013) Cell, 154, 1380–1389.
А. и др. (2013) Cell, 154, 1380–1389. - Qi, L.S., et al. (2013) Cell, 152, 1173–1183.
- Гасюнас Г. и др. (2012) Proc. Натл. акад. науч. США, 109, E2579–2586.
- Maede, M.L., et al. (2013) Нац. Методы, 10, 977–979.
- Гилберт, Л.А., и др. (2013) Cell, 154, 442–451.
- Hu, J., et al. (2014) Nucleic Acids Res. doi:10.1093/nar/gku109.
- Перес-Пинера П. и др. (2013) Нац. Методы, 10, 239–242.
- Чен Б. и др. (2013) Cell, 155, 1479–1491.
- Seung, W., et al. (2014) Genome Res. 24, 132–141.
- Миллер, Дж. К. и др. (2011). Нац. Биотехнолог. 29, 143–148.
- Муссолино, К., и др. (2011). Рез. нуклеиновых кислот. 39, 9283–9293.
- Maeder, M.L., et al. (2008) Мол. Сотовый, 31, 294–301.
- Hwang, W.Y., et al. (2013) PLoS One, 8:e68708.
- Фэн З.
 и др. (2013) Cell Res. 23, 1229–1232.
и др. (2013) Cell Res. 23, 1229–1232. - Мали, П. и др. (2013) Наука, 339, 823–826.
- Чжоу Дж. и др. (2014) FEBS J. doi:10.1111/febs.12735.
- Fu, Y., et al. (2013) Нац. Биотехнолог. 31, 822–826.
- Фу, Ю. и др. (2014) Nat Biotechnol. doi: 10.1038/nbt.2808.
- Хсу, П.Д., и др. (2013) Нац. Биотехнолог. 31, 827–832.
- Сандер, Дж. Д. и др. (2007) Nucleic Acids Res. 35, W599-605.
- Sander, J.D., et al (2010) Nucleic Acids Res. 38, W462–468.
- Пайн М.Е. и др. (2015) Appl Environ Microbiol 81:5103–5114.
- О, Дж. Х. и др. (2014) Nucleic Acids Res 42:e131.
- Цзян В. и др. (2013) Nat Biotechnol 31:233–239.
- Хай Т. и др. (2014) Cell Res. doi: 10.1038/cr.2014.11.
- Guo, X., et al. (2014) Развитие, 141, 707–714.
- ДиКарло, Дж. Э. и др.
 (2013) Nucleic Acids Res. 41, 4336–4343.
(2013) Nucleic Acids Res. 41, 4336–4343. - Грац С.Дж. и др. (2014) Genetics, doi:10.1534/genetics.113.160713.
- Ню, Ю. и др. (2014) Cell, 156, 836–843.
- Ян Д. и др. (2014) J. Mol. Клеточная биол. 6, 97-99.
- Ма, Ю. и др. (2014) Cell Res. 24, 122–125.
- Машико Д. и др. (2014) Дев. Разница в росте. 56, 122–129.
- Грац С.Дж. и др. (2013) Муха, 249.
- Мали, П. и др. (2013) Нац. Биотехнолог. 31, 833–838.
- Ченг А.В. и др. (2013) Сотовый рез. 23, 1163–1171.
- Koike-Yusa, H., et al. (2013) Нац. Биотехнолог. doi: 10.1038/nbt.2800.
- Сандер, Дж.Д., и Юнг, Дж.К. (2014) Nat Biotechnol. doi:10.1038/nbt.2842.
Из выражений NEB, выпуск I, 2014 г.
Статья Алекса Рейса, доктора философии, Bitesize Bio
Бретон Хорнблауэр, доктор философии, Бретт Робб, доктор философии.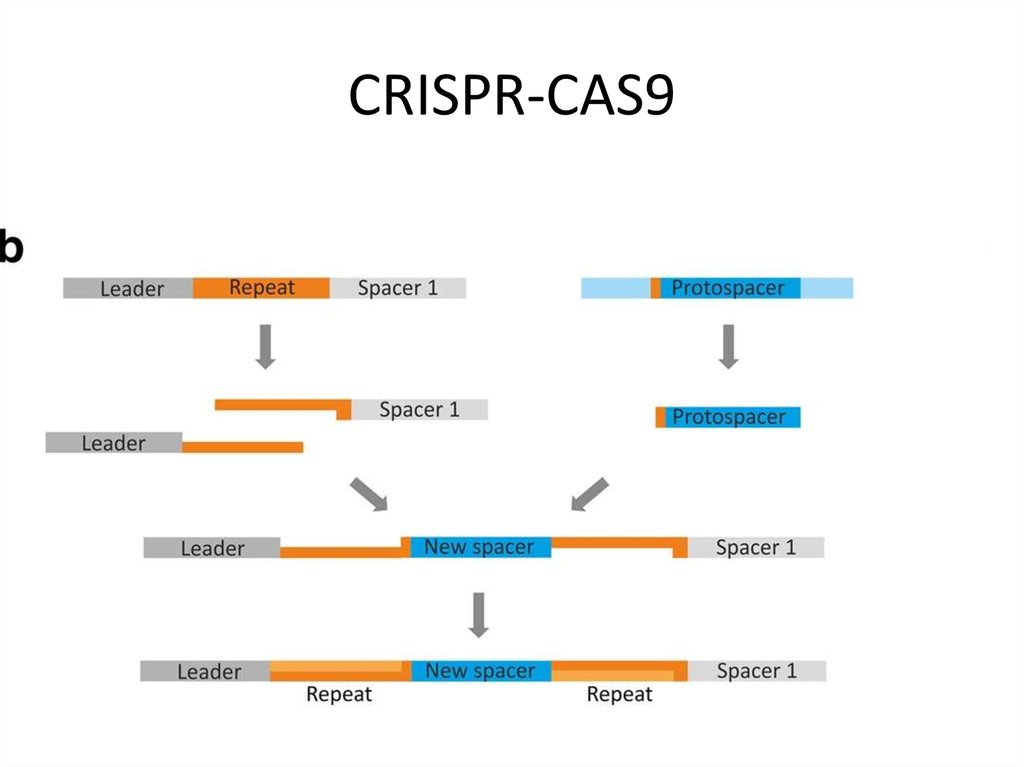 и Джордж Церцинис, доктор философии, New England
и Джордж Церцинис, доктор философии, New England
Biolabs, Inc.
Введение в CRISPR/Cas9система
Мы используем файлы cookie, чтобы улучшить ваш просмотр и предоставить полезный контент. Ознакомьтесь с нашей политикой в отношении файлов cookie.
Принимать
- Логин пользователя
- Регистр
- Просмотр корзины (0)
- Логин пользователя
- Регистр
- Просмотр корзины (0)
Товары
Обучение
Документация
Дом
›
Учебные центры
›
Генная функция
›
Редактирование генов
›
Инструменты и информация для редактирования генов
›
Введение в CRISPR/Cas9система
Редактирование генов
Средство поиска продуктов редактирования генов
Инструменты и информация для редактирования генов
инструменты дизайна sgRNA
Инструменты для успешного редактирования генома CRISPR/Cas9
Плакаты редактирования генов
Данные клиентов для продуктов Guide-it
Как разработать последовательности sgRNA
Введение в систему CRISPR/Cas9
Редактирование генов CD3+ Т-клеток и CD34+ HSC
Нокауты CRISPR/Cas9
Нокины CRISPR/Cas9
Полногеномный скрининг
Создание и проверка SNP
Способы доставки CRISPR/Cas9
Cre рекомбиназа
Новые продукты
Связаться с отделом продаж
Мощный метод конструирования интересующего гена
Хотя недавно разработанные программируемые инструменты редактирования, такие как нуклеазы цинковых пальцев и эффекторные нуклеазы, подобные активаторам транскрипции, значительно расширили возможности точной модификации генома, эти методы имеют ограничения. CRISPR (сгруппированные короткие палиндромные повторы с регулярными интервалами)/Cas9Технология представляет собой значительное улучшение по сравнению с другими инструментами редактирования генома следующего поколения, достигая нового уровня нацеливания, эффективности и простоты использования. Система CRISPR/Cas9 позволяет нацеливать геномные сайты практически на любой организм.
CRISPR (сгруппированные короткие палиндромные повторы с регулярными интервалами)/Cas9Технология представляет собой значительное улучшение по сравнению с другими инструментами редактирования генома следующего поколения, достигая нового уровня нацеливания, эффективности и простоты использования. Система CRISPR/Cas9 позволяет нацеливать геномные сайты практически на любой организм.
Система CRISPR/Cas типа II представляет собой прокариотическую систему адаптивного иммунного ответа, которая использует некодирующие РНК, чтобы направлять нуклеазу Cas9 для индукции сайт-специфического расщепления ДНК. Это повреждение ДНК восстанавливается с помощью механизмов репарации клеточной ДНК, либо через негомологичный путь репарации ДНК с соединением концов (NHEJ), либо через путь репарации, направленной на гомологию (HDR).
Система CRISPR/Cas9 была использована для создания простого РНК-программируемого метода редактирования генома в клетках млекопитающих, который можно использовать для создания нокаутов генов (путем вставки/удаления) или нокинов (через HDR). Чтобы создать нарушения в генах (рис. 1), создается одна направляющая РНК (sgRNA), которая направляет нуклеазу Cas9 в определенное место в геноме. Вызванные Cas9 двухцепочечные разрывы восстанавливаются с помощью пути репарации ДНК NHEJ. Репарация подвержена ошибкам, поэтому могут быть введены вставки и делеции (INDEL), которые могут нарушить функцию гена.
Чтобы создать нарушения в генах (рис. 1), создается одна направляющая РНК (sgRNA), которая направляет нуклеазу Cas9 в определенное место в геноме. Вызванные Cas9 двухцепочечные разрывы восстанавливаются с помощью пути репарации ДНК NHEJ. Репарация подвержена ошибкам, поэтому могут быть введены вставки и делеции (INDEL), которые могут нарушить функцию гена.
Принцип разрушения генов, опосредованного CRISPR/Cas9. Одна направляющая РНК (sgRNA), состоящая из последовательности crРНК, специфичной для ДНК-мишени, и последовательности tracrRNA, взаимодействующей с белком Cas9 (1), связывается с рекомбинантной формой белка Cas9, обладающей ДНК-эндонуклеазной активностью ( 2). Образовавшийся комплекс вызывает специфическое расщепление двухцепочечной ДНК (3). Сайт расщепления будет восстанавливаться с помощью негомологичного соединения концов (NHEJ) пути репарации ДНК, подверженного ошибкам процесса, который может привести к вставкам/делециям (INDEL), которые могут нарушить функцию гена (4).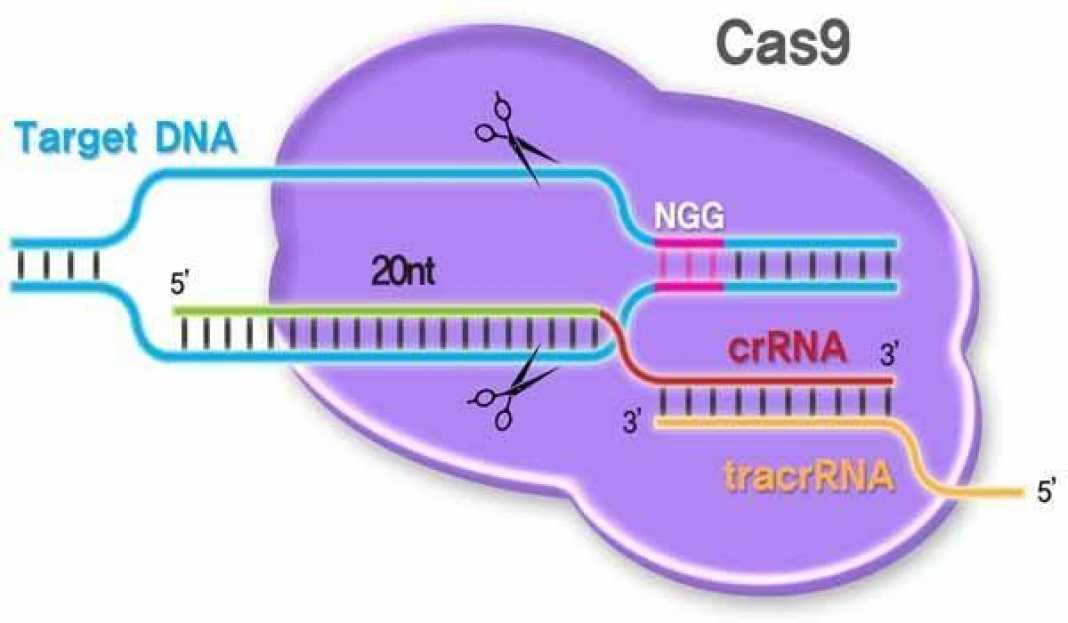
Технология
CRISPR/Cas9 произвела революцию в редактировании генома, обеспечив недостижимый ранее уровень геномного нацеливания, эффективности и простоты. Продукты Guide-it еще больше повышают удобство использования системы CRISPR/Cas9, предоставляя оптимизированный метод для:
- Производство sgRNA in vitro
- Проверка эффективности расщепления sgRNAs
- Мониторинг эффективности редактирования генома в культивируемых клетках
Информация CRISPR/Cas9
Ознакомьтесь с коллекцией инструментов проектирования sgRNA для экспериментов по редактированию генома на основе Cas9.
Узнайте, как создавать последовательности sgRNA для успешного редактирования генов.
Обзор системы CRISPR/Cas9 для редактирования генома.
Обзор инструментов, доступных для каждого этапа успешного рабочего процесса редактирования генома.
Доставка Cas9 и sgRNA в клетки млекопитающих с использованием различных инновационных инструментов.
Узнайте о простом анализе для чувствительного обнаружения однонуклеотидных замен в популяциях отредактированных или клонированных клеток.
Узнайте о компонентах и рабочем процессе Guide-it CRISPR/Cas9 Gesicle Production System.
900:30 Посмотрите этот веб-семинар, чтобы узнать, как легко проводить полногеномный скрининг лентивирусной sgRNA.
Посмотрите вебинар о том, как правильно выбрать HDR-шаблон для экспериментов.
Получите ответы на часто задаваемые вопросы и просмотрите видео, объясняющее ферментативный анализ.
Takara Bio USA, Inc.
США/Канада: +1 800 662 2566 • Азиатско-Тихоокеанский регион: +1 650,919.

 Помимо улучшения пищевых качеств, такими инструментами легко наделять культуры устойчивостью к вредителям и химикалиям, а животных — избавлять от нежелательных генов. Например, недавно в свиных клетках инактивировали эндогенных ретровирусов (не то чтобы здоровья свиньи ради, а держа в голове планы по трансплантации ее органов человеку) [18];
Помимо улучшения пищевых качеств, такими инструментами легко наделять культуры устойчивостью к вредителям и химикалиям, а животных — избавлять от нежелательных генов. Например, недавно в свиных клетках инактивировали эндогенных ретровирусов (не то чтобы здоровья свиньи ради, а держа в голове планы по трансплантации ее органов человеку) [18]; Новые или оптимизированные ферментные системы бактерий и грибов, получаемые так легко и дешево, — предел мечтаний технологов из целого ряда отраслей промышленности. Но даже простое встраивание в промышленно важные штаммы бактерий систем CRISPR-Cas с заданными свойствами может защитить их от бактериофагов и нежелательных плазмид.
Новые или оптимизированные ферментные системы бактерий и грибов, получаемые так легко и дешево, — предел мечтаний технологов из целого ряда отраслей промышленности. Но даже простое встраивание в промышленно важные штаммы бактерий систем CRISPR-Cas с заданными свойствами может защитить их от бактериофагов и нежелательных плазмид. Cпособность к разрезанию CRISPR-кассеты для встраивания новых спейсеров, скорее всего, вторичное функциональное приобретение Cas1. Исходно он резал типичные рекомбинационные/репарационные интермедиаты и пока что не разучился это делать. Поэтому экспрессия генов cas повышает устойчивость некоторых бактерий к радиации, а отключение CRISPR-системы ведет к повышению их чувствительности к повреждающим ДНК факторам и нарушению расхождения хромосом;
Cпособность к разрезанию CRISPR-кассеты для встраивания новых спейсеров, скорее всего, вторичное функциональное приобретение Cas1. Исходно он резал типичные рекомбинационные/репарационные интермедиаты и пока что не разучился это делать. Поэтому экспрессия генов cas повышает устойчивость некоторых бактерий к радиации, а отключение CRISPR-системы ведет к повышению их чувствительности к повреждающим ДНК факторам и нарушению расхождения хромосом; Функция эта по сути тоже иммунная, но экстремальная. Предполагают, что в избранных CRISPR-системах один из Cas-белков может служить «токсином», а его партнер — «антитоксином», и когда в клетку проникает фаг, «токсин» высвобождается и начинает крушить любую РНК. Но у «замирающей» при этом клетки есть время лихорадочно насобирать спейсеры. Если это не получается, то разбушевавшийся Cas-токсин доводит клетку до самоубийства. Доказать этот принцип пока не удалось [7]. Зато подобный драматический финал мы уже где-то видели: именно так поступает система VI типа. Такой исход фаговой инвазии называется абортивной инфекцией. Для клетки плохо, для популяции — хорошо…
Функция эта по сути тоже иммунная, но экстремальная. Предполагают, что в избранных CRISPR-системах один из Cas-белков может служить «токсином», а его партнер — «антитоксином», и когда в клетку проникает фаг, «токсин» высвобождается и начинает крушить любую РНК. Но у «замирающей» при этом клетки есть время лихорадочно насобирать спейсеры. Если это не получается, то разбушевавшийся Cas-токсин доводит клетку до самоубийства. Доказать этот принцип пока не удалось [7]. Зато подобный драматический финал мы уже где-то видели: именно так поступает система VI типа. Такой исход фаговой инвазии называется абортивной инфекцией. Для клетки плохо, для популяции — хорошо… Mol Microbiol. 43, 1565-1575;
Mol Microbiol. 43, 1565-1575; Lander. (2016). The Heroes of CRISPR. Cell. 164, 18-28;
Lander. (2016). The Heroes of CRISPR. Cell. 164, 18-28; (2015). Application of CRISPR/Cas9 genome editing to the study and treatment of disease. Arch Toxicol. 89, 1023-1034;
(2015). Application of CRISPR/Cas9 genome editing to the study and treatment of disease. Arch Toxicol. 89, 1023-1034; Translational Research. 168, 15-21;
Translational Research. 168, 15-21;
 Это означает, что, по крайней мере теоретически, направляющая РНК будет связываться только с последовательностью-мишенью и никакими другими областями генома.
Это означает, что, по крайней мере теоретически, направляющая РНК будет связываться только с последовательностью-мишенью и никакими другими областями генома.

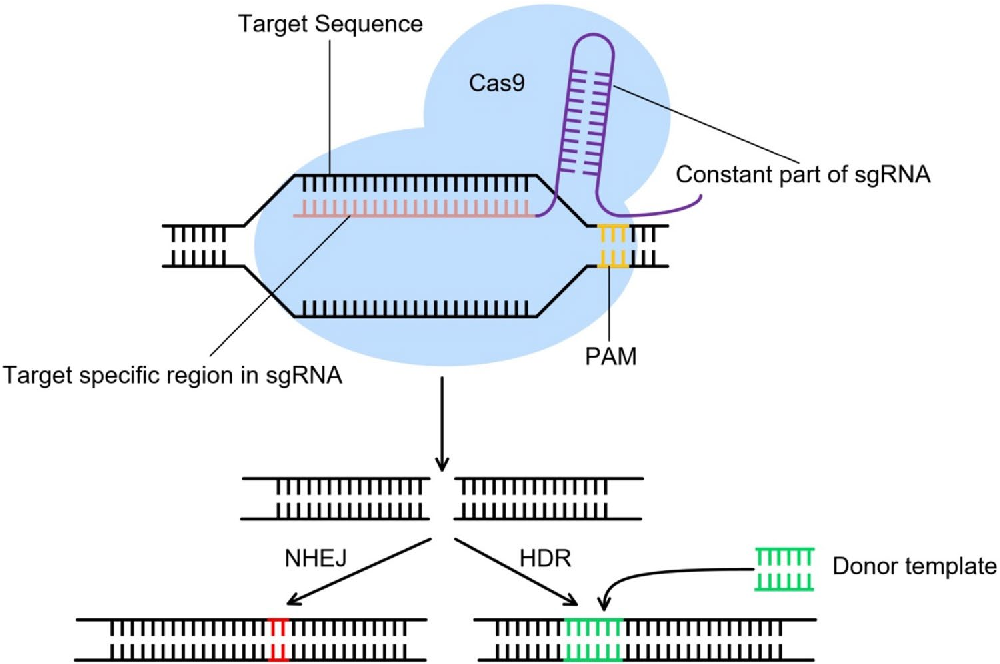
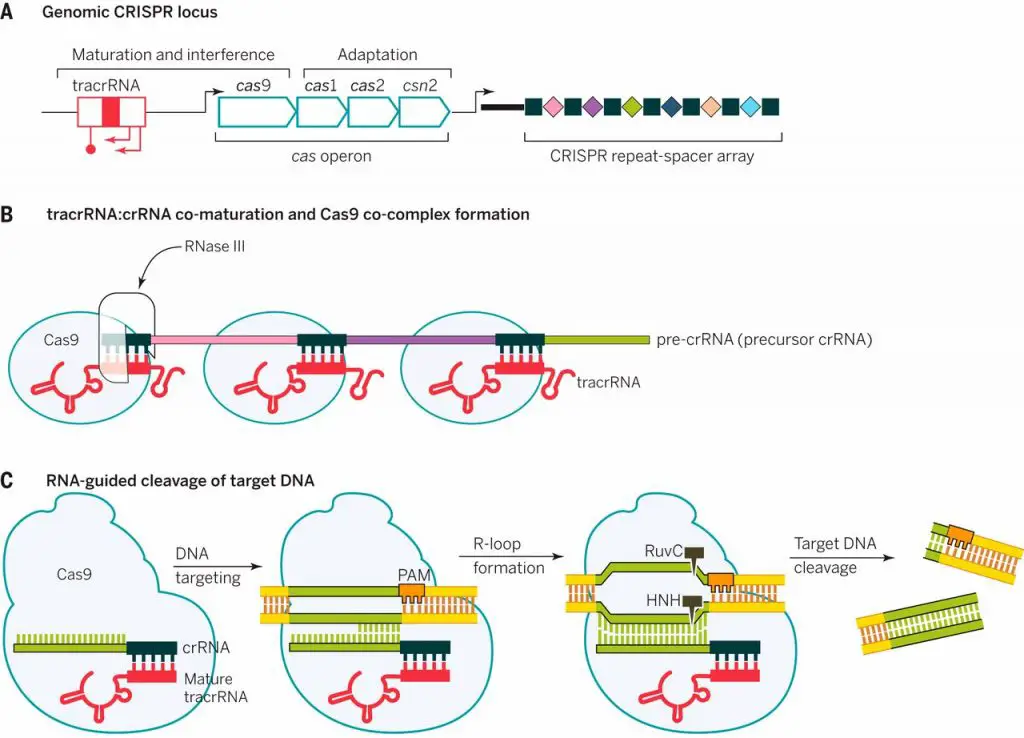


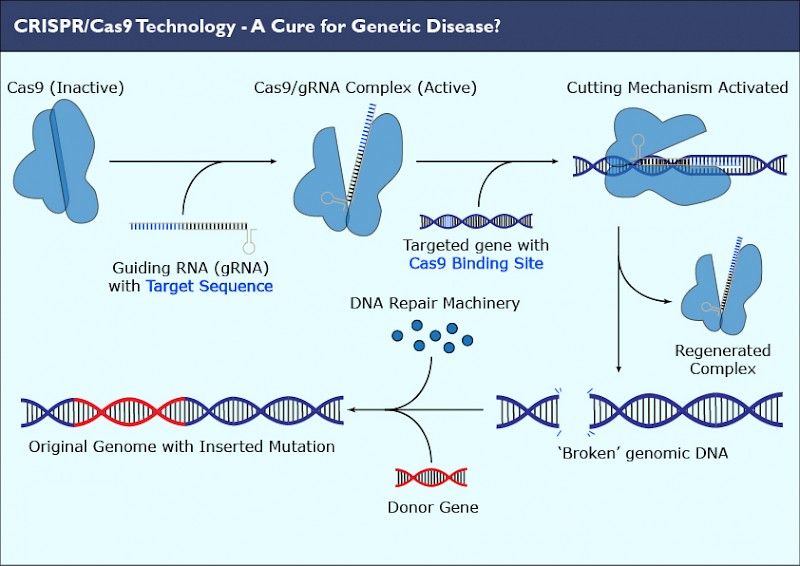 А. и др. (2013) Cell, 154, 1380–1389.
А. и др. (2013) Cell, 154, 1380–1389.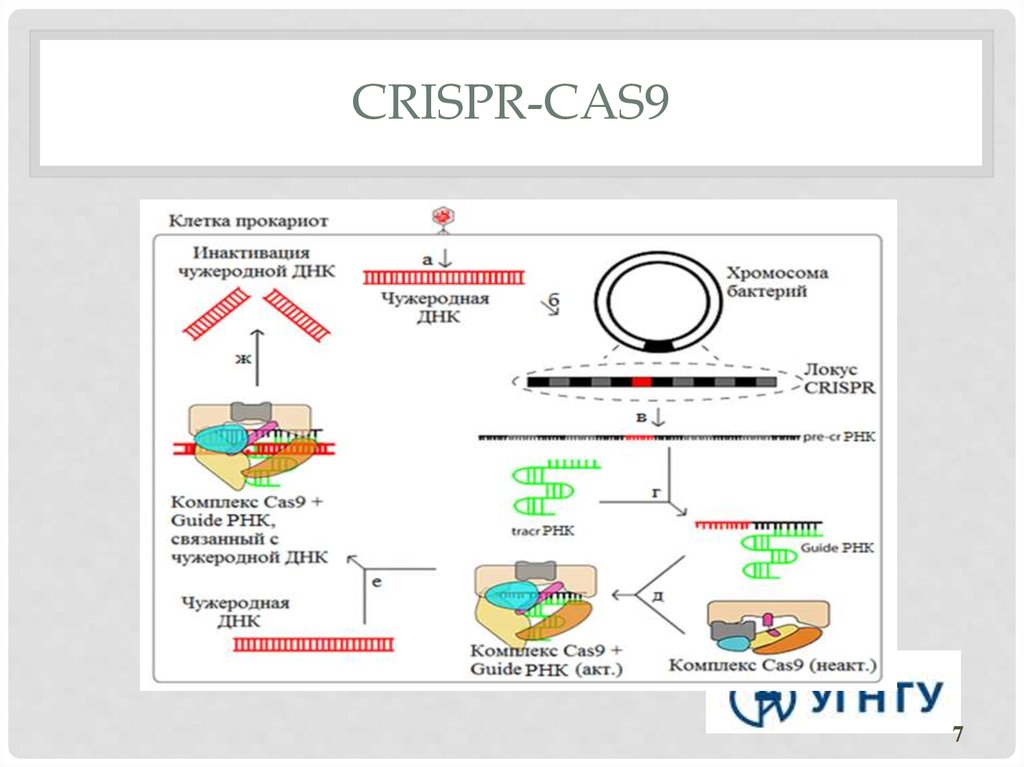 и др. (2013) Cell Res. 23, 1229–1232.
и др. (2013) Cell Res. 23, 1229–1232. (2013) Nucleic Acids Res. 41, 4336–4343.
(2013) Nucleic Acids Res. 41, 4336–4343.