Содержание
ДНК анализ «Секвенирование генома» — полная расшифровка всей ДНК
Секвенирование генома — это эксклюзивная услуга, которая предоставит Вам и Вашему врачу полную информацию обо всех Ваших генах. В отличие от услуги «Мой геном» и аналогичных предложений других компаний, когда при анализе используется технология ДНК-чипов, позволяющая определить не более 1 млн. нуклеотидов, секвенирование всего генома позволяет узнать все 3 миллиарда нуклеотидов, составляющих Вашу ДНК
Секвенирование генома человека играет все более важную роль в формировании персонализированной системы здравоохранения. Этот метод предоставляет персональную генетическую информацию, необходимую для эффективного диагностирования и целенаправленного лечения болезне.
В настоящее время полногемное секвенирование становится не только научной, но и медицинской задачей, в связи с чем, многие известные люди уже воспользовались этой услугой.
Вот что говорил о геноме Джеймс Уотсон, лауреат Нобелевской премии за открытие структуры ДНК, чей геном был расшифрован в 2007 году (это стоило $1. 5 миллиона): «Однажды я сидел за рулем Bentley — это хорошая машина, — говорит — Что бы я предпочёл — узнать полную последовательность моего генома или владеть Bentley? Пожалуй, я бы выбрал полную последовательность генома, потому что она может показать гены, определяющие предрасположенность к заболеваниям. С генами я передам моим детям и информацию о предрасположенности к ряду заболеваний, хотя, быть может, и не заболею ими сам. Что на самом деле необходимо человечеству, так это «геном-Chevrolet», который по средствам каждому».
5 миллиона): «Однажды я сидел за рулем Bentley — это хорошая машина, — говорит — Что бы я предпочёл — узнать полную последовательность моего генома или владеть Bentley? Пожалуй, я бы выбрал полную последовательность генома, потому что она может показать гены, определяющие предрасположенность к заболеваниям. С генами я передам моим детям и информацию о предрасположенности к ряду заболеваний, хотя, быть может, и не заболею ими сам. Что на самом деле необходимо человечеству, так это «геном-Chevrolet», который по средствам каждому».
Если Вы заинтересовались услугой по полногеномному секвенированию, Вам необходимо получить консультацию врача-генетика, чтобы понять, позволит ли данная услуга получить исчерпывающие ответы на интересующие Вас вопросы.
После секвенирования генома доктор обсудит с Вами полученную информацию и составит индивидуальный план действий, который может включать в себя дополнительные медицинские исследования, направленные на профилактику или превентивное лечение заболеваний, а также сохранение Вашей молодости и продление жизни.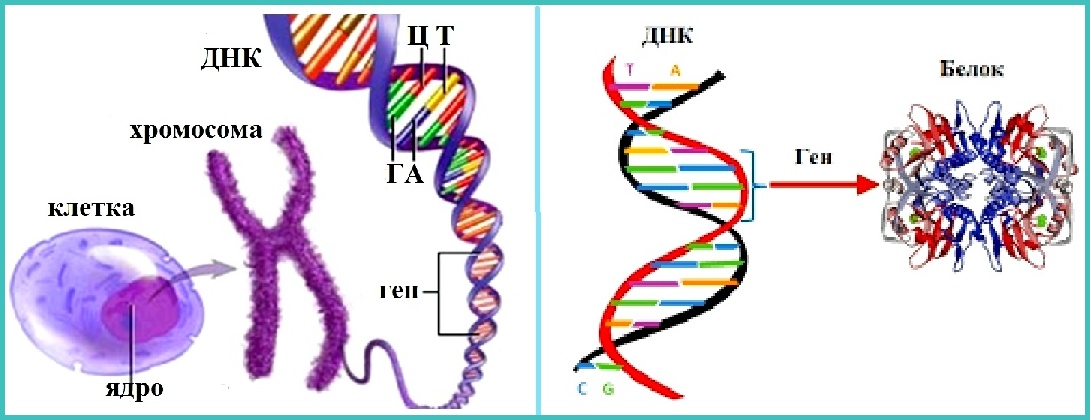
На протяжении всего времени анализа Вашего генома, которое составит около 4 месяцев, на любые Ваши вопросы будут отвечать ваш персональный менеджер и врач-генетик. В результате анализа вы получите книгу, в которой будет рассказано о Ваших предрасположенностях и генетических особенностях, а также трехлетнюю подписку на он-лайн обновления Genotek, которые позволят Вам узнавать все самые новые сведения о Вашем геноме, открытые учеными со всего мира. Также вы получите флеш-накопитель с исходными данными о Вашем геноме, которые могут потребоваться при обращении за медицинской помощью.
В 2008 году Дэн Стоическу, миллиардер, живущий в Швейцарии, стал одним из первых людей в мире, кто заказал расшифровку своего генома. Это стоило ему $350 000. «Я предпочел потратить деньги на расшифровку генома, нежели покупку автомобиля или самолета,» — говорит Стоикеску, которому в 2008 году исполнилось 56. Через два года после выхода на пенсию он задумался о том, какие генетические риски несет его генетический код. Стоикеску говорит, что будет ежедневно смотреть за обновлением его генетической информации, ведь каждый день появляются все новые сведения о связи многих заболеваний и его генов.
Стоикеску говорит, что будет ежедневно смотреть за обновлением его генетической информации, ведь каждый день появляются все новые сведения о связи многих заболеваний и его генов.
По единогласному мнению ученых, секвенирование генома в течение ближайших лет останется эксклюзивной услугой премиум-класса. Сегодня в мире лишь несколько компаний могут предложить своим клиентам полное секвенирование генома, в том числе компания Genotek.
Если вы хотите узнать более полную информацию, цены, или уже готовы сделать секвенирование генома – обращайтесь по указанной ссылке: https://www.genotek.ru/
Ученые поставили финальную точку в расшифровке генома человека
Исследователи представили эталонный образец расшифровки полной последовательности генома человека, который может быть использован в медико-генетических лабораториях. Сравнивая имеющиеся образцы с эталоном, можно искать мутации в генах, вызывающие различные заболевания.
В международную научную группу вошли представители Научно-технологического университета «Сириус» под руководством директора Научного центра генетики и наук о жизни, наставника двух ведущих программ магистратуры университета Евгения Рогаева. Опубликованные в журнале Science научные статьи
Опубликованные в журнале Science научные статьи
можно считать финальной точкой в вопросе расшифровки генома человека. Фундаментальное достижение ученых теперь доступно для использования всем членам мирового научного сообщества.
Спустя 20 лет после первой публикации научному консорциуму удалось впервые описать полную последовательность 22 аутосом человека, а также X-хромосомы «от кончика до кончика». За последнее время многие научные группы секвенировали и выполняли сборку генома человека, однако данные сборки были неполными, и около 8% не удавалось представить в виде единой последовательности.
Теперь ученые смогли правильно заполнить пробелы и восстановить полную картину. Это стало возможным благодаря усовершенствованию методов секвенирования с длинными прочтениями, которые позволили охватить повторяющиеся регионы, что ранее было основным препятствием для создания полной сборки.
Над расшифровкой трудились ученые из более чем 30 организаций, в том числе из России, где в работе принимали участие ведущие специалисты в области биологии центромер, основной вклад в которую принадлежит кандидату биологических наук Ивану Александрову.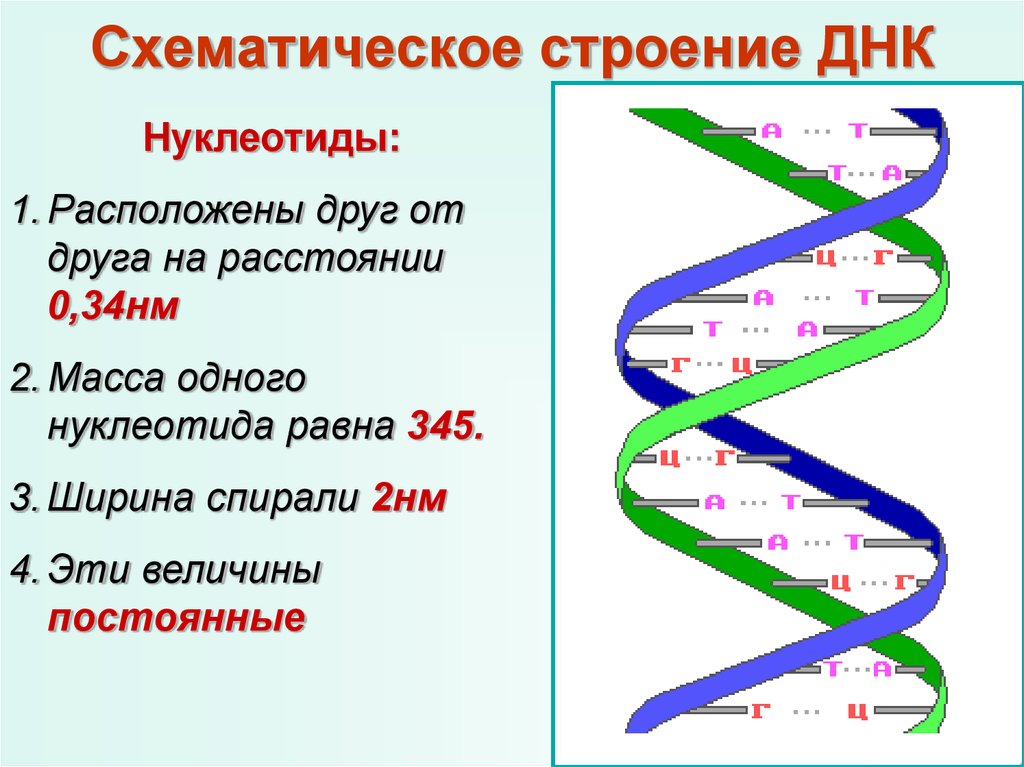
Анализ различных аспектов новой сборки проводился несколькими научными группами, каждая из которых подготовила свои результаты к публикации. Ученые университета «Сириус» внесли свой вклад в две публикации — результаты работы по секвенированию и сборке генома образца CHM13 и анализу центромерных повторов в хромосомах. Кроме того, они отвечали за валидацию сборки центромерных областей хромосом ранее целиком не декодированных участков в геноме человека — эти материалы вошли в статью «The complete sequence of a human genome». А для статьи «Complete genomic and epigenetic maps of human centromeres» научная группа подготовила детальные аннотации центромерных и перицентромерных регионов, а также провела всесторонний анализ повторов в геноме.
Представленная расшифровка генома — это эталонный образец, который может быть использован в медико-генетических лабораториях. Сравнивая имеющиеся образцы с эталоном, можно искать мутации в генах, вызывающие различные заболевания или, напротив, повышающие приспособляемость организмов. Такая работа, например, уже ведется учеными «Сириуса» в рамках проекта по изучению геномов долгожителей России.
Такая работа, например, уже ведется учеными «Сириуса» в рамках проекта по изучению геномов долгожителей России.
«Получая информацию о геноме, мы читаем короткие фрагменты, а затем сравниваем их с эталоном, который был расшифрован. Таким образом мы ищем варианты, которые есть у долгожителей и которые способны уберечь их от распространенных возрастных заболеваний. Так, возможно, в будущем ученым удастся блокировать гены, отвечающие за многие болезни», — пояснил Евгений Рогаев.
«Нам удалось показать, что центромеры человека эволюционируют посредством «расширения слоев», то есть более новые последовательности, расширяясь из активной центромеры хромосомы, оттесняют более старые к флангам, которые со временем накапливают мутации и частично сокращаются. В результате мы можем проследить эволюцию центромерных повторов, поскольку наш геном содержит остатки центромер древних приматов», — объясняет Лев Уральский, научный сотрудник Центра генетики и наук о жизни.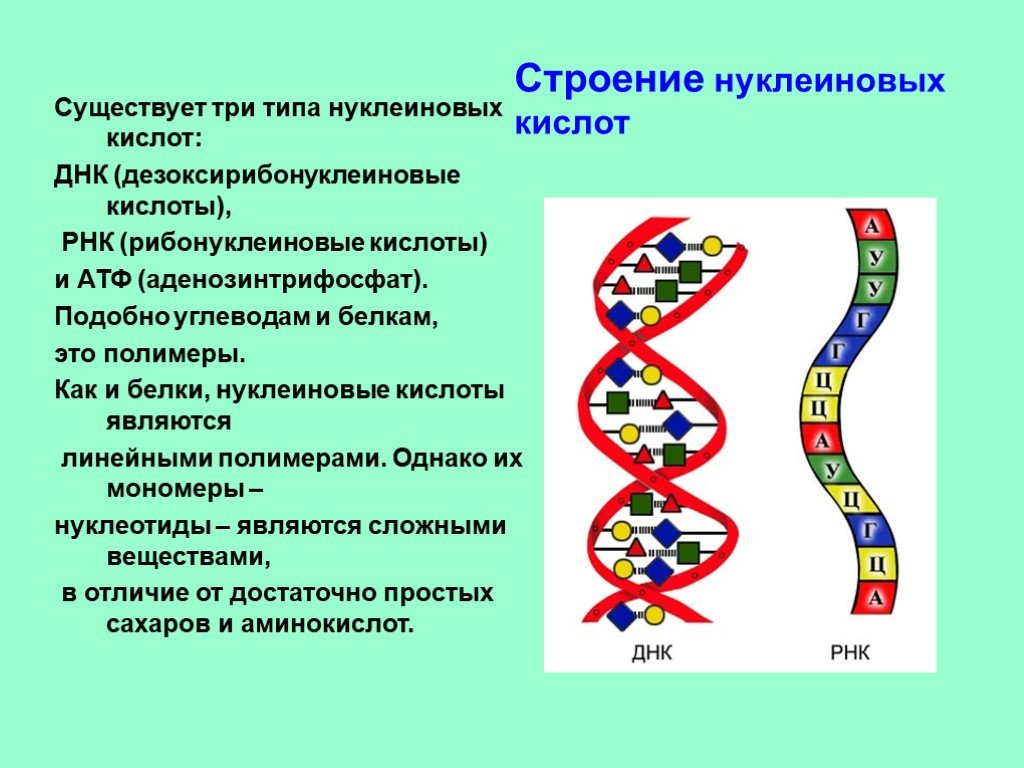
Кроме того, ученые «Сириуса» продолжают исследования в области расшифровки генома древних образцов в рамках проекта по генетической истории древнего населения Русской равнины при поддержке Министерства науки и высшего образования Российской Федерации.
Совместно с ведущими археологическими институтами России была собрана уникальная база ДНК древних людей — более 200 образцов, работа по секвенированию которых впервые проводится в нашей стране. На основе этих исследований можно реконструировать историю человечества, опираясь на науку и факты, а не на письменные источники, которые часто бывают необъективными. Также, по словам специалистов, исследование ДНК древних людей и сравнение с геномом современного человека позволит предсказать появление в будущем новых заболеваний и даст возможность избежать их.
DeCoDe: дизайн вырожденных кодонов для полных библиотек ДНК, кодирующих белок
. 2020 1 июня; 36 (11): 3357-3364.
doi: 10.1093/биоинформатика/btaa162.
Тайлер С Шимко
1
, Полли М. Фордайс
1
2
3
4
, Ярон Оренштейн
5
Принадлежности
- 1 Кафедра генетики.
- 2 Кафедра биоинженерии.
- 3 Stanford ChEM-H, Стэнфордский университет, Стэнфорд, Калифорния 94305, США.
- 4 Биохаб Чана Цукерберга, Сан-Франциско, Калифорния 94158, США.

- 5 Факультет электротехники и вычислительной техники Университета Бен-Гуриона в Негеве, Беэр-Шева 8410501, Израиль.
PMID:
32176271
PMCID:
PMC7267834
DOI:
10.1093/биоинформатика/btaa162
Бесплатная статья ЧВК
Тайлер С. Шимко и др.
Биоинформатика.
.
Бесплатная статья ЧВК
. 2020 1 июня; 36 (11): 3357-3364.
doi: 10.1093/биоинформатика/btaa162.
Авторы
Тайлер С Шимко
1
, Полли М. Фордайс
1
2
3
4
, Ярон Оренштейн
5
Принадлежности
- 1 Кафедра генетики.
- 2 Кафедра биоинженерии.
- 3 Stanford ChEM-H, Стэнфордский университет, Стэнфорд, Калифорния 94305, США.
- 4 Биохаб Чана Цукерберга, Сан-Франциско, Калифорния 94158, США.

- 5 Факультет электротехники и вычислительной техники Университета Бен-Гуриона в Негеве, Беэр-Шева 8410501, Израиль.
PMID:
32176271
PMCID:
PMC7267834
DOI:
10.1093/биоинформатика/btaa162
Абстрактный
Мотивация:
Скрининг белка с высокой пропускной способностью является критически важным методом анализа и разработки функции белка. Библиотеки для этих анализов могут быть созданы с помощью ряда средств, включая целенаправленный или случайный мутагенез матричной белковой последовательности или прямой синтез ДНК. Однако методы конструирования мутагенных библиотек часто дают гораздо больше нефункциональных, чем функциональных вариантов, и, несмотря на успехи в крупномасштабном синтезе ДНК, индивидуальный синтез каждой желаемой ДНК-матрицы часто чрезмерно дорог. Следовательно, многие библиотеки для скрининга белков полагаются на использование вырожденных кодонов (DC), смесей оснований ДНК, включенных в определенные положения во время синтеза ДНК, для создания весьма разнообразных пулов вариантов белков всего за несколько недорогих реакций синтеза. Однако выбор DC для наборов последовательностей, ковариирующихся в нескольких позициях, резко увеличивает сложность разработки библиотеки DC и приводит к созданию многих нежелательных вариантов, которые могут быстро превзойти возможности скрининга.
Однако методы конструирования мутагенных библиотек часто дают гораздо больше нефункциональных, чем функциональных вариантов, и, несмотря на успехи в крупномасштабном синтезе ДНК, индивидуальный синтез каждой желаемой ДНК-матрицы часто чрезмерно дорог. Следовательно, многие библиотеки для скрининга белков полагаются на использование вырожденных кодонов (DC), смесей оснований ДНК, включенных в определенные положения во время синтеза ДНК, для создания весьма разнообразных пулов вариантов белков всего за несколько недорогих реакций синтеза. Однако выбор DC для наборов последовательностей, ковариирующихся в нескольких позициях, резко увеличивает сложность разработки библиотеки DC и приводит к созданию многих нежелательных вариантов, которые могут быстро превзойти возможности скрининга.
Полученные результаты:
Мы представляем новый алгоритм полной оптимизации библиотеки DC, дизайн вырожденных кодонов (DeCoDe), основанный на целочисленном линейном программировании. DeCoDe значительно превосходит современные алгоритмы оптимизации DC и хорошо масштабируется до более чем сотни белков, имеющих сложные модели ковариации (например, полученная в лаборатории линия avGFP). Более того, DeCoDe, насколько нам известно, является первым алгоритмом проектирования DC с возможностью кодирования белковых библиотек смешанной длины. Мы ожидаем, что DeCoDe будет широко полезен для решения множества проблем создания библиотек, начиная от попыток инженерии белков, которые используют взаимную информацию, и заканчивая реконструкцией состояний белков предков.
DeCoDe значительно превосходит современные алгоритмы оптимизации DC и хорошо масштабируется до более чем сотни белков, имеющих сложные модели ковариации (например, полученная в лаборатории линия avGFP). Более того, DeCoDe, насколько нам известно, является первым алгоритмом проектирования DC с возможностью кодирования белковых библиотек смешанной длины. Мы ожидаем, что DeCoDe будет широко полезен для решения множества проблем создания библиотек, начиная от попыток инженерии белков, которые используют взаимную информацию, и заканчивая реконструкцией состояний белков предков.
Наличие и реализация:
github.com/OrensteinLab/DeCoDe.
Контакт:
Дополнительная информация:
Дополнительные данные доступны на веб-сайте Bioinformatics Online.
© Автор(ы), 2020. Опубликовано Oxford University Press. Все права защищены. Для разрешений, пожалуйста, по электронной почте: [email protected].
Опубликовано Oxford University Press. Все права защищены. Для разрешений, пожалуйста, по электронной почте: [email protected].
Цифры
Рис. 1.
Примеры ковариационных структур в…
Рис. 1.
Примеры ковариационных структур в наборах целевых белков (верхний ряд). Для каждой ковариации…
Рисунок 1.
Примеры ковариационных структур в наборах целевых белков (верхний ряд). Для каждого шаблона ковариации была создана оптимальная библиотека с ограничением общего размера в шесть последовательностей ДНК. Аминокислотные последовательности, закодированные этой оптимизированной библиотекой, показаны (нижний ряд), а качество соответствующей библиотеки, измеряемое количеством охватываемых целевых последовательностей, показано под каждой оптимизированной библиотекой. ( A ) Комплект с двумя независимо изменяющимися положениями. Библиотека DC, созданная для этого набора последовательностей, кодирует все шесть мишеней. ( B ) Набор с двумя совпадающими позициями. Библиотека DC, созданная для этого набора последовательностей, кодирует только четыре из шести мишеней. Позиция третьей переменной не является вырожденной в оптимальной библиотеке. ( C ) Набор с несколькими ковариантными позициями и мутацией indel. Библиотека DC, созданная для этого набора последовательностей, кодирует только три из шести целей. При добавлении второй подбиблиотеки мутация indel обрабатывается шаблонами разной длины, а общее покрытие составляет пять целей 9.0003
( A ) Комплект с двумя независимо изменяющимися положениями. Библиотека DC, созданная для этого набора последовательностей, кодирует все шесть мишеней. ( B ) Набор с двумя совпадающими позициями. Библиотека DC, созданная для этого набора последовательностей, кодирует только четыре из шести мишеней. Позиция третьей переменной не является вырожденной в оптимальной библиотеке. ( C ) Набор с несколькими ковариантными позициями и мутацией indel. Библиотека DC, созданная для этого набора последовательностей, кодирует только три из шести целей. При добавлении второй подбиблиотеки мутация indel обрабатывается шаблонами разной длины, а общее покрытие составляет пять целей 9.0003
Рис. 2.
Производительность DeCoDe и SwiftLib на…
Рис. 2.
Производительность DeCoDe и SwiftLib в тесте 1XBI с использованием четырех подбиблиотек.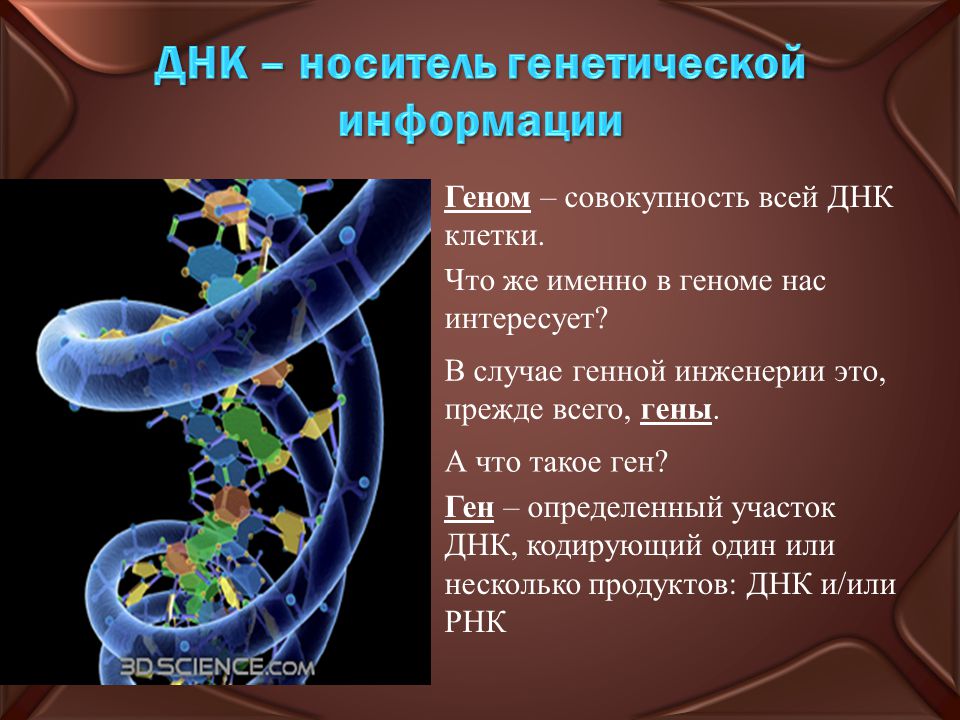 ( А…
( А…
Рис. 2.
Производительность
DeCoDe и SwiftLib в тесте 1XBI с использованием четырех подбиблиотек. ( A ) Общее количество полноразмерных целевых последовательностей, охваченных каждой оптимизированной библиотекой. ( B, C ) Сравнение полной ковариации высшего порядка. Каждая точка представляет собой долю от общего (B) или уникального (C) целевого k -мера, охваченного оптимизированными библиотеками, созданными каждым методом. Значения k обозначены цветом каждой точки
Рис. 3.
Сравнение полноразмерной мишени и…
Рис. 3.
Сравнение полноразмерного целевого и k -мерного покрытия DeCoDe и SwiftLib на…
Рис.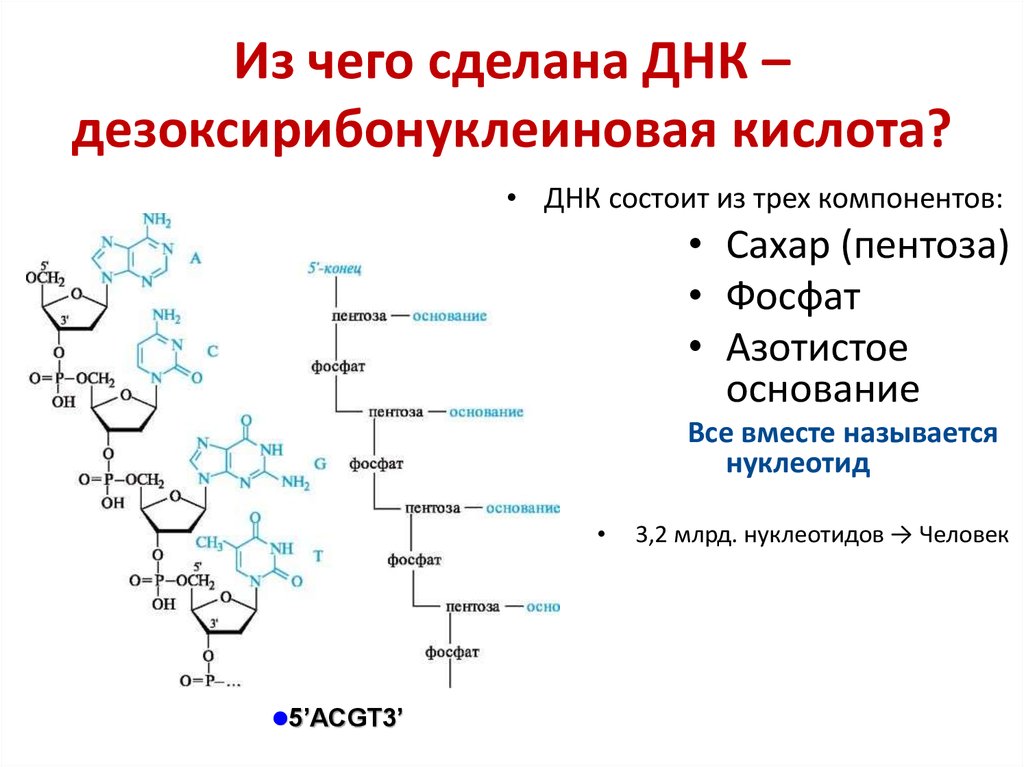 3.
3.
Сравнение охвата полной длины мишени и k -mer с помощью DeCoDe и SwiftLib на наборе целевых последовательностей из 94 белков-мишеней, происходящих от avGFP, длиной 239 аминокислот, где 73 позиции являются вариабельными между белками. ( A, B ) Всего охвачено целевые показатели для библиотек, состоящих из одной подбиблиотеки и двух подбиблиотек соответственно. ( C, D ) Охвачена доля от общего числа и уникальных целевых тыс. -меров. Различные размеры библиотек обозначаются цветом, а использование одной или двух подбиблиотек обозначается формой
.
См. это изображение и информацию об авторских правах в PMC
Похожие статьи
CoLiDe: Инструмент для создания комбинаторной библиотеки для исследования пространства белковых последовательностей.
Третьяченко В., Ворачек В.
 , Соучек Р., Фудзисима К., Глухова К.
, Соучек Р., Фудзисима К., Глухова К.
Третьяченко В, и др.
Биоинформатика. 2021 1 мая; 37 (4): 482-489. doi: 10.1093/биоинформатика/btaa804.
Биоинформатика. 2021.PMID: 32956450
Бесплатная статья ЧВК.SwiftLib: быстрая оптимизация библиотеки вырожденных кодонов с помощью динамического программирования.
Джейкобс ТМ, Юмерефенди Х, Кульман Б, Ливер-Фэй А.
Джейкобс ТМ и др.
Нуклеиновые Кислоты Res. 2015 11 марта; 43 (5): e34. дои: 10.1093/нар/гку1323. Epub 2014 24 декабря.
Нуклеиновые Кислоты Res. 2015.PMID: 25539925
Бесплатная статья ЧВК.Автоматизированный дизайн библиотек вырожденных кодонов.
Мена Массачусетс, Догерти PS.
Мена М.А. и соавт.
Белок Eng Des Sel. 2005 Декабрь; 18 (12): 559-61. doi: 10.1093/белок/gzi061. Epub 2005 20 октября.
doi: 10.1093/белок/gzi061. Epub 2005 20 октября.
Белок Eng Des Sel. 2005.PMID: 16239261
Оптимизация комбинаторного мутагенеза.
Паркер А.С., Грисволд К.Е., Бейли-Келлог К.
Паркер А.С. и др.
J Компьютерная биология. 2011 ноябрь;18(11):1743-56. doi: 10.1089/cmb.2011.0152. Epub 2011, 16 сентября.
J Компьютерная биология. 2011.PMID: 21923411
Бесплатная статья ЧВК.Смешанные олигонуклеотиды для случайного мутагенеза: лучший способ их получения.
Аруначалам Т.С., Вихерт С., Аппель Б., Мюллер С.
Аруначалам Т.С. и др.
Орг Биомол Хим. 2012 28 июня; 10 (24): 4641-50. дои: 10.1039/c2ob25328c. Epub 2012 2 мая.
Орг Биомол Хим. 2012.PMID: 22552713
Обзор.
Посмотреть все похожие статьи
Цитируется
uPIC-M: Эффективная и масштабируемая подготовка библиотек клональных одиночных мутантов для высокопроизводительной биохимии белков.

Аппель М.Дж., Лонгвелл С.А., Морри М., Нефф Н., Хершлаг Д., Фордайс П.М.
Аппель М.Дж. и соавт.
АСУ Омега. 2021 2 ноября; 6(45):30542-30554. doi: 10.1021/acsomega.1c04180. Электронная коллекция 2021 16 ноября.
АСУ Омега. 2021.PMID: 34805683
Бесплатная статья ЧВК.CoLiDe: Инструмент для создания комбинаторной библиотеки для исследования пространства белковых последовательностей.
Третьяченко В., Ворачек В., Соучек Р., Фудзисима К., Глухова К.
Третьяченко В, и др.
Биоинформатика. 2021 1 мая; 37(4):482-489. doi: 10.1093/биоинформатика/btaa804.
Биоинформатика. 2021.PMID: 32956450
Бесплатная статья ЧВК.
Типы публикаций
термины MeSH
вещества
Грантовая поддержка
- DP2 GM123641/GM/NIGMS NIH HHS/США
Новый компьютерный алгоритм расшифровывает самые сокровенные секреты ДНК; может помочь найти связи между генами и болезнями — ScienceDaily
Геном – это руководство по эксплуатации организма. Он содержит необработанную информацию — в форме ДНК — которая определяет все, от того, ходит ли животное на четырех или двух ногах, до потенциального риска заболевания. Но это руководство написано на языке биологии, поэтому разобраться во всем, что в нем зашифровано, оказалось непросто. Теперь исследователи Колумбийского университета разработали вычислительный инструмент, который проливает свет на самые трудные для перевода сегменты генома. Имея в руках этот инструмент, ученые могут приблизиться к пониманию того, как ДНК управляет всем, от роста и развития до старения и болезней.
Он содержит необработанную информацию — в форме ДНК — которая определяет все, от того, ходит ли животное на четырех или двух ногах, до потенциального риска заболевания. Но это руководство написано на языке биологии, поэтому разобраться во всем, что в нем зашифровано, оказалось непросто. Теперь исследователи Колумбийского университета разработали вычислительный инструмент, который проливает свет на самые трудные для перевода сегменты генома. Имея в руках этот инструмент, ученые могут приблизиться к пониманию того, как ДНК управляет всем, от роста и развития до старения и болезней.
Исследователи недавно опубликовали свои выводы в журнале Proceedings of the National Academy of Sciences .
«Геномы даже простейших организмов, таких как плодовая муха, содержат 120 миллионов букв ДНК, большую часть которых еще предстоит расшифровать, потому что сигналы, которые она дает, были слишком тонкими для существующих инструментов», — сказал Ричард Манн. , доктор философии, главный исследователь Колумбийского института поведения мозга и мозга Мортимера Б.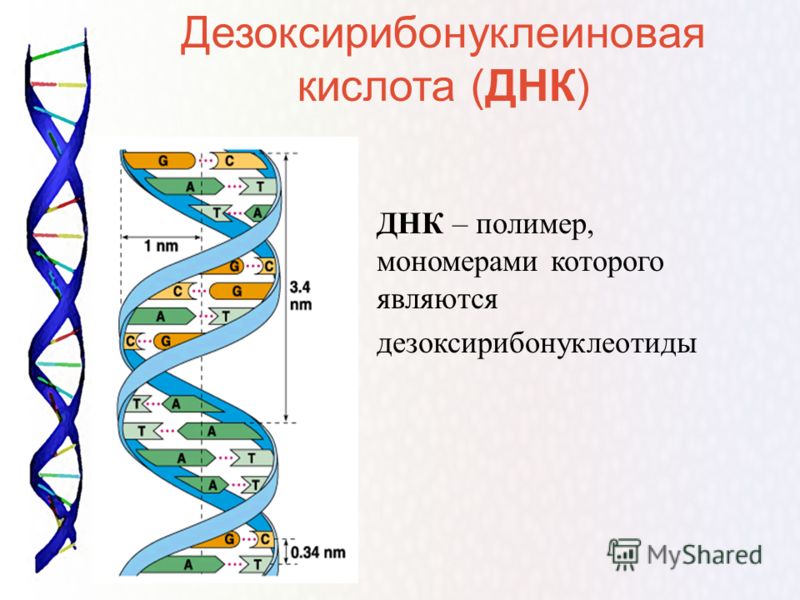 Цукермана и старший автор статьи. «Но наш новый алгоритм позволяет нам пролистывать эти миллионы строк генетического кода и улавливать даже самые слабые сигналы, что дает гораздо более полную картину того, что кодирует ДНК».
Цукермана и старший автор статьи. «Но наш новый алгоритм позволяет нам пролистывать эти миллионы строк генетического кода и улавливать даже самые слабые сигналы, что дает гораздо более полную картину того, что кодирует ДНК».
Генетики долго искали способы расшифровать тайны, скрытые в ДНК. Одна из таких загадок связана с особенно распространенным классом генов, известных как Hox-гены.
«Гены Hox являются главными архитекторами тела; они управляют некоторыми из самых ранних и наиболее важных аспектов роста и дифференцировки, например, где у развивающегося эмбриона должны располагаться голова и конечности», — сказал доктор Манн, который также является Хиггинс, профессор биохимии и молекулярной биофизики (в системной биологии) Медицинского центра Ирвинга Колумбийского университета. «Гены Hox делают это, производя белки, называемые транскрипционными факторами, которые связываются с последовательностями ДНК, чтобы включать или выключать большие группы генов; это похоже на переключение тысяч переключателей в точно правильном порядке».
Но десятилетия исследований Hox-генов выявили парадокс: даже несмотря на то, что каждый отдельный Hox-ген контролировал разные особенности роста, все Hox-факторы транскрипции прочно и явно связывались с одним и тем же набором легко идентифицируемых последовательностей ДНК.
В 2015 году д-р Манн и его команда обнаружили, что факторы транскрипции Hox также связываются во многих других местах — просто более дискретно в так называемых «сайтах с низким сродством». Ученые полагали, что эти сайты связывания с низким сродством являются ключевыми факторами транскрипции Hox, способными управлять одним аспектом развития по сравнению с другим. Оставалась проблема, как расшифровать эти участки из генома.
Чтобы решить эту проблему, д-р Манн и его лаборатория объединили усилия с лабораторией Хармена Буссемейкера, доктора философии, профессора кафедры биологических наук и системной биологии Колумбийского университета и эксперта по построению вычислительных моделей генетической активности.
Несколько лет назад две лаборатории разработали метод генетического секвенирования под названием SELEX-seq для систематической характеристики всех сайтов связывания Hox. Но у их подхода все еще были ограничения: он требовал, чтобы один и тот же фрагмент ДНК секвенировался снова и снова. С каждым новым раундом открывалось больше кусочков головоломки, но информация об этих критических сайтах связывания с низким сродством оставалась скрытой.
«Это было похоже на то, как если бы один и тот же абзац прогонялся через Google Translate несколько раз, но в итоге точно переводилось только десять процентов слов», — сказал д-р Манн.
Чтобы решить эту проблему, доктор Буссемейкер и его команда разработали новый сложный компьютерный алгоритм, который впервые смог объяснить поведение всех последовательностей ДНК в эксперименте SELEX-seq. Они назвали этот алгоритм No Read Left Behind или NRLB.
«Проще говоря, NRLB позволяет нам охватить весь спектр сайтов связывания — от самого высокого до самого низкого сродства — с гораздо большей степенью чувствительности и точности, чем любой существующий метод, включая современный.


 , Соучек Р., Фудзисима К., Глухова К.
, Соучек Р., Фудзисима К., Глухова К. doi: 10.1093/белок/gzi061. Epub 2005 20 октября.
doi: 10.1093/белок/gzi061. Epub 2005 20 октября.